Introdução Link para o cabeçalho
Recentemente, o Hospital Albert Einstein compartilhou um conjunto de dados com observações de 5644 pacientes. Os dados possuem 111 características relacionadas a exames médicos, como sangue, urina e muito mais. A tarefa que me propus a resolver foi classificar os pacientes testados em dois grupos, aqueles que posssuem e aqueles que não possuem coronavírus. Farei isso a partir a partir dos resultados dos exames laboratoriais, sem utilizar informação alguma sobre o diagnóstico realizado em relação à contaminação do vírus.
Caso a técnica utilizada neste posto não tenha ficado muito clara, recomendo a leitura do meu post Tutorial: Como Fazer o Seu Primeiro Projeto de Data Science, que mostra com mais detalhes como ajustar o modelo random forest a um conjunto de dados.
Os dados utilizados aqui, bem como o código utilizado nesta análise, estão disponíveis em meu github.
Análise Exploratória dos Dados Link para o cabeçalho
O primeiro passo em qualquer análise de dados é a análise exploratória. É possível gerar ideias e insights apenas olhando para os dados plotados. Vou começar minha análise exploratória carregando alguns pacotes no R:
library(tidyverse)
theme_set(theme_bw())
library(naniar)
library(readxl)
library(caret)
library(reshape2)
library(GGally)
library(mice)
Com os pacotes carregados, vou ler os dados e realizar pequenas correções neles. Cada passo a seguir foi explicado com um comentário dentro do próprio código.
dataset <- read_excel(path = "data/dataset.xlsx")
# fix column names
names(dataset) <- make.names(names(dataset), unique = TRUE)
#################################
### exploratory data analysis ###
#################################
# first look at the dataset
glimpse(dataset)
## Rows: 5,644
## Columns: 111
## $ Patient.ID <chr> "44477f75e8169d2…
## $ Patient.age.quantile <dbl> 13, 17, 8, 5, 15…
## $ SARS.Cov.2.exam.result <chr> "negative", "neg…
## $ Patient.addmited.to.regular.ward..1.yes..0.no. <dbl> 0, 0, 0, 0, 0, 0…
## $ Patient.addmited.to.semi.intensive.unit..1.yes..0.no. <dbl> 0, 0, 0, 0, 0, 0…
## $ Patient.addmited.to.intensive.care.unit..1.yes..0.no. <dbl> 0, 0, 0, 0, 0, 0…
## $ Hematocrit <dbl> NA, 0.2365154, N…
## $ Hemoglobin <dbl> NA, -0.02234027,…
## $ Platelets <dbl> NA, -0.51741302,…
## $ Mean.platelet.volume <dbl> NA, 0.01067657, …
## $ Red.blood.Cells <dbl> NA, 0.1020042, N…
## $ Lymphocytes <dbl> NA, 0.318365753,…
## $ Mean.corpuscular.hemoglobin.concentration..MCHC. <dbl> NA, -0.9507903, …
## $ Leukocytes <dbl> NA, -0.09461035,…
## $ Basophils <dbl> NA, -0.22376651,…
## $ Mean.corpuscular.hemoglobin..MCH. <dbl> NA, -0.29226932,…
## $ Eosinophils <dbl> NA, 1.4821582, N…
## $ Mean.corpuscular.volume..MCV. <dbl> NA, 0.1661924, N…
## $ Monocytes <dbl> NA, 0.35754666, …
## $ Red.blood.cell.distribution.width..RDW. <dbl> NA, -0.6250727, …
## $ Serum.Glucose <dbl> NA, -0.1406481, …
## $ Respiratory.Syncytial.Virus <chr> NA, "not_detecte…
## $ Influenza.A <chr> NA, "not_detecte…
## $ Influenza.B <chr> NA, "not_detecte…
## $ Parainfluenza.1 <chr> NA, "not_detecte…
## $ CoronavirusNL63 <chr> NA, "not_detecte…
## $ Rhinovirus.Enterovirus <chr> NA, "detected", …
## $ Mycoplasma.pneumoniae <lgl> NA, NA, NA, NA, …
## $ Coronavirus.HKU1 <chr> NA, "not_detecte…
## $ Parainfluenza.3 <chr> NA, "not_detecte…
## $ Chlamydophila.pneumoniae <chr> NA, "not_detecte…
## $ Adenovirus <chr> NA, "not_detecte…
## $ Parainfluenza.4 <chr> NA, "not_detecte…
## $ Coronavirus229E <chr> NA, "not_detecte…
## $ CoronavirusOC43 <chr> NA, "not_detecte…
## $ Inf.A.H1N1.2009 <chr> NA, "not_detecte…
## $ Bordetella.pertussis <chr> NA, "not_detecte…
## $ Metapneumovirus <chr> NA, "not_detecte…
## $ Parainfluenza.2 <chr> NA, "not_detecte…
## $ Neutrophils <dbl> NA, -0.6190860, …
## $ Urea <dbl> NA, 1.19805908, …
## $ Proteina.C.reativa.mg.dL <dbl> NA, -0.1478949, …
## $ Creatinine <dbl> NA, 2.0899284, N…
## $ Potassium <dbl> NA, -0.3057871, …
## $ Sodium <dbl> NA, 0.8625116, N…
## $ Influenza.B..rapid.test <chr> NA, "negative", …
## $ Influenza.A..rapid.test <chr> NA, "negative", …
## $ Alanine.transaminase <dbl> NA, NA, NA, NA, …
## $ Aspartate.transaminase <dbl> NA, NA, NA, NA, …
## $ Gamma.glutamyltransferase. <dbl> NA, NA, NA, NA, …
## $ Total.Bilirubin <dbl> NA, NA, NA, NA, …
## $ Direct.Bilirubin <dbl> NA, NA, NA, NA, …
## $ Indirect.Bilirubin <dbl> NA, NA, NA, NA, …
## $ Alkaline.phosphatase <dbl> NA, NA, NA, NA, …
## $ Ionized.calcium. <dbl> NA, NA, NA, NA, …
## $ Strepto.A <chr> NA, NA, NA, NA, …
## $ Magnesium <dbl> NA, NA, NA, NA, …
## $ pCO2..venous.blood.gas.analysis. <dbl> NA, NA, NA, NA, …
## $ Hb.saturation..venous.blood.gas.analysis. <dbl> NA, NA, NA, NA, …
## $ Base.excess..venous.blood.gas.analysis. <dbl> NA, NA, NA, NA, …
## $ pO2..venous.blood.gas.analysis. <dbl> NA, NA, NA, NA, …
## $ Fio2..venous.blood.gas.analysis. <lgl> NA, NA, NA, NA, …
## $ Total.CO2..venous.blood.gas.analysis. <dbl> NA, NA, NA, NA, …
## $ pH..venous.blood.gas.analysis. <dbl> NA, NA, NA, NA, …
## $ HCO3..venous.blood.gas.analysis. <dbl> NA, NA, NA, NA, …
## $ Rods.. <dbl> NA, NA, NA, NA, …
## $ Segmented <dbl> NA, NA, NA, NA, …
## $ Promyelocytes <dbl> NA, NA, NA, NA, …
## $ Metamyelocytes <dbl> NA, NA, NA, NA, …
## $ Myelocytes <dbl> NA, NA, NA, NA, …
## $ Myeloblasts <dbl> NA, NA, NA, NA, …
## $ Urine...Esterase <chr> NA, NA, NA, NA, …
## $ Urine...Aspect <chr> NA, NA, NA, NA, …
## $ Urine...pH <chr> NA, NA, NA, NA, …
## $ Urine...Hemoglobin <chr> NA, NA, NA, NA, …
## $ Urine...Bile.pigments <chr> NA, NA, NA, NA, …
## $ Urine...Ketone.Bodies <chr> NA, NA, NA, NA, …
## $ Urine...Nitrite <chr> NA, NA, NA, NA, …
## $ Urine...Density <dbl> NA, NA, NA, NA, …
## $ Urine...Urobilinogen <chr> NA, NA, NA, NA, …
## $ Urine...Protein <chr> NA, NA, NA, NA, …
## $ Urine...Sugar <lgl> NA, NA, NA, NA, …
## $ Urine...Leukocytes <chr> NA, NA, NA, NA, …
## $ Urine...Crystals <chr> NA, NA, NA, NA, …
## $ Urine...Red.blood.cells <dbl> NA, NA, NA, NA, …
## $ Urine...Hyaline.cylinders <chr> NA, NA, NA, NA, …
## $ Urine...Granular.cylinders <chr> NA, NA, NA, NA, …
## $ Urine...Yeasts <chr> NA, NA, NA, NA, …
## $ Urine...Color <chr> NA, NA, NA, NA, …
## $ Partial.thromboplastin.time..PTT.. <lgl> NA, NA, NA, NA, …
## $ Relationship..Patient.Normal. <dbl> NA, NA, NA, NA, …
## $ International.normalized.ratio..INR. <dbl> NA, NA, NA, NA, …
## $ Lactic.Dehydrogenase <dbl> NA, NA, NA, NA, …
## $ Prothrombin.time..PT...Activity <lgl> NA, NA, NA, NA, …
## $ Vitamin.B12 <dbl> NA, NA, NA, NA, …
## $ Creatine.phosphokinase..CPK.. <dbl> NA, NA, NA, NA, …
## $ Ferritin <dbl> NA, NA, NA, NA, …
## $ Arterial.Lactic.Acid <dbl> NA, NA, NA, NA, …
## $ Lipase.dosage <lgl> NA, NA, NA, NA, …
## $ D.Dimer <lgl> NA, NA, NA, NA, …
## $ Albumin <dbl> NA, NA, NA, NA, …
## $ Hb.saturation..arterial.blood.gases. <dbl> NA, NA, NA, NA, …
## $ pCO2..arterial.blood.gas.analysis. <dbl> NA, NA, NA, NA, …
## $ Base.excess..arterial.blood.gas.analysis. <dbl> NA, NA, NA, NA, …
## $ pH..arterial.blood.gas.analysis. <dbl> NA, NA, NA, NA, …
## $ Total.CO2..arterial.blood.gas.analysis. <dbl> NA, NA, NA, NA, …
## $ HCO3..arterial.blood.gas.analysis. <dbl> NA, NA, NA, NA, …
## $ pO2..arterial.blood.gas.analysis. <dbl> NA, NA, NA, NA, …
## $ Arteiral.Fio2 <dbl> NA, NA, NA, NA, …
## $ Phosphor <dbl> NA, NA, NA, NA, …
## $ ctO2..arterial.blood.gas.analysis. <dbl> NA, NA, NA, NA, …
# remove columns that won't help on diagnosis
dataset_clean <- dataset %>%
select(-Patient.ID,
-Patient.addmited.to.regular.ward..1.yes..0.no.,
-Patient.addmited.to.semi.intensive.unit..1.yes..0.no.,
-Patient.addmited.to.intensive.care.unit..1.yes..0.no.)
# convert level Urine...Leukocytes <1000 to 1000
dataset_clean$Urine...Leukocytes[dataset_clean$`Urine...Leukocytes` == "<1000"] <- 1000
dataset_clean$`Urine...Leukocytes` <- as.numeric(dataset_clean$`Urine...Leukocytes`)
# fix Urine...pH
dataset_clean$`Urine...pH`[dataset_clean$`Urine...pH` == "Não Realizado"] <- NA
dataset_clean$`Urine...pH` <- as.numeric(dataset_clean$`Urine...pH`)
# Urine...Hemoglobin
dataset_clean$`Urine...Hemoglobin`[dataset_clean$`Urine...Hemoglobin` == "not_done"] <- NA
# Urine...Aspect
dataset_clean$`Urine...Aspect` <- factor(dataset_clean$`Urine...Aspect`,
levels = c("clear", "lightly_cloudy", "cloudy", "altered_coloring"))
# Strepto A
dataset_clean$`Strepto.A`[dataset_clean$`Strepto.A` == "not_done"] <- NA
# transform character to factor
dataset_clean_num <- dataset_clean %>%
select_if(is.numeric)
dataset_clean_cat <- dataset_clean %>%
select_if(negate(is.numeric)) %>%
mutate_all(as.factor)
dataset_clean <- base::cbind(dataset_clean_num, dataset_clean_cat)
# fix factor levels
# sort(sapply(dataset_clean[,sapply(dataset_clean, is.factor)], nlevels))
Com os dados lidos e processados, eu dou uma olhada nos dados faltantes:
# let's take a look on missing data
missing_values <- dataset_clean %>%
gather(key = "key", value = "val") %>%
mutate(is.missing = is.na(val)) %>%
group_by(key, is.missing) %>%
summarise(num.missing = n()) %>%
filter(is.missing == TRUE) %>%
select(-is.missing) %>%
ungroup() %>%
mutate(key = reorder(key, -num.missing)) %>%
arrange(desc(num.missing)) %>%
print(n = Inf)
## Warning: attributes are not identical across measure variables; they will be
## dropped
## `summarise()` has grouped output by 'key'. You can override using the `.groups`
## argument.
## # A tibble: 105 × 2
## key num.missing
## <fct> <int>
## 1 D.Dimer 5644
## 2 Mycoplasma.pneumoniae 5644
## 3 Partial.thromboplastin.time..PTT.. 5644
## 4 Prothrombin.time..PT...Activity 5644
## 5 Urine...Sugar 5644
## 6 Fio2..venous.blood.gas.analysis. 5643
## 7 Urine...Nitrite 5643
## 8 Vitamin.B12 5641
## 9 Lipase.dosage 5636
## 10 Albumin 5631
## 11 Arteiral.Fio2 5624
## 12 Phosphor 5624
## 13 Ferritin 5621
## 14 Arterial.Lactic.Acid 5617
## 15 Base.excess..arterial.blood.gas.analysis. 5617
## 16 HCO3..arterial.blood.gas.analysis. 5617
## 17 Hb.saturation..arterial.blood.gases. 5617
## 18 Total.CO2..arterial.blood.gas.analysis. 5617
## 19 ctO2..arterial.blood.gas.analysis. 5617
## 20 pCO2..arterial.blood.gas.analysis. 5617
## 21 pH..arterial.blood.gas.analysis. 5617
## 22 pO2..arterial.blood.gas.analysis. 5617
## 23 Magnesium 5604
## 24 Ionized.calcium. 5594
## 25 Urine...Ketone.Bodies 5587
## 26 Urine...Esterase 5584
## 27 Urine...Protein 5584
## 28 Urine...Hyaline.cylinders 5577
## 29 Urine...Granular.cylinders 5575
## 30 Urine...Hemoglobin 5575
## 31 Urine...Urobilinogen 5575
## 32 Urine...pH 5575
## 33 Urine...Aspect 5574
## 34 Urine...Bile.pigments 5574
## 35 Urine...Color 5574
## 36 Urine...Crystals 5574
## 37 Urine...Density 5574
## 38 Urine...Leukocytes 5574
## 39 Urine...Red.blood.cells 5574
## 40 Urine...Yeasts 5574
## 41 Relationship..Patient.Normal. 5553
## 42 Metamyelocytes 5547
## 43 Myeloblasts 5547
## 44 Myelocytes 5547
## 45 Promyelocytes 5547
## 46 Rods.. 5547
## 47 Segmented 5547
## 48 Lactic.Dehydrogenase 5543
## 49 Creatine.phosphokinase..CPK.. 5540
## 50 International.normalized.ratio..INR. 5511
## 51 Base.excess..venous.blood.gas.analysis. 5508
## 52 HCO3..venous.blood.gas.analysis. 5508
## 53 Hb.saturation..venous.blood.gas.analysis. 5508
## 54 Total.CO2..venous.blood.gas.analysis. 5508
## 55 pCO2..venous.blood.gas.analysis. 5508
## 56 pH..venous.blood.gas.analysis. 5508
## 57 pO2..venous.blood.gas.analysis. 5508
## 58 Alkaline.phosphatase 5500
## 59 Gamma.glutamyltransferase. 5491
## 60 Direct.Bilirubin 5462
## 61 Indirect.Bilirubin 5462
## 62 Total.Bilirubin 5462
## 63 Serum.Glucose 5436
## 64 Alanine.transaminase 5419
## 65 Aspartate.transaminase 5418
## 66 Strepto.A 5313
## 67 Sodium 5274
## 68 Potassium 5273
## 69 Urea 5247
## 70 Creatinine 5220
## 71 Proteina.C.reativa.mg.dL 5138
## 72 Neutrophils 5131
## 73 Mean.platelet.volume 5045
## 74 Monocytes 5043
## 75 Basophils 5042
## 76 Eosinophils 5042
## 77 Leukocytes 5042
## 78 Lymphocytes 5042
## 79 Mean.corpuscular.hemoglobin..MCH. 5042
## 80 Mean.corpuscular.hemoglobin.concentration..MCHC. 5042
## 81 Mean.corpuscular.volume..MCV. 5042
## 82 Platelets 5042
## 83 Red.blood.Cells 5042
## 84 Red.blood.cell.distribution.width..RDW. 5042
## 85 Hematocrit 5041
## 86 Hemoglobin 5041
## 87 Influenza.A..rapid.test 4824
## 88 Influenza.B..rapid.test 4824
## 89 Adenovirus 4292
## 90 Bordetella.pertussis 4292
## 91 Chlamydophila.pneumoniae 4292
## 92 Coronavirus.HKU1 4292
## 93 Coronavirus229E 4292
## 94 CoronavirusNL63 4292
## 95 CoronavirusOC43 4292
## 96 Inf.A.H1N1.2009 4292
## 97 Metapneumovirus 4292
## 98 Parainfluenza.1 4292
## 99 Parainfluenza.2 4292
## 100 Parainfluenza.3 4292
## 101 Parainfluenza.4 4292
## 102 Rhinovirus.Enterovirus 4292
## 103 Influenza.A 4290
## 104 Influenza.B 4290
## 105 Respiratory.Syncytial.Virus 4290
missing_values %>%
ggplot() +
geom_bar(aes(x = key, y = 100*num.missing/dim(dataset_clean)[1]), stat = "identity") +
labs(x = "Variable", y="Percent of missing values") +
theme(axis.text.x = element_text(angle = 45, hjust = 1, size = 8))
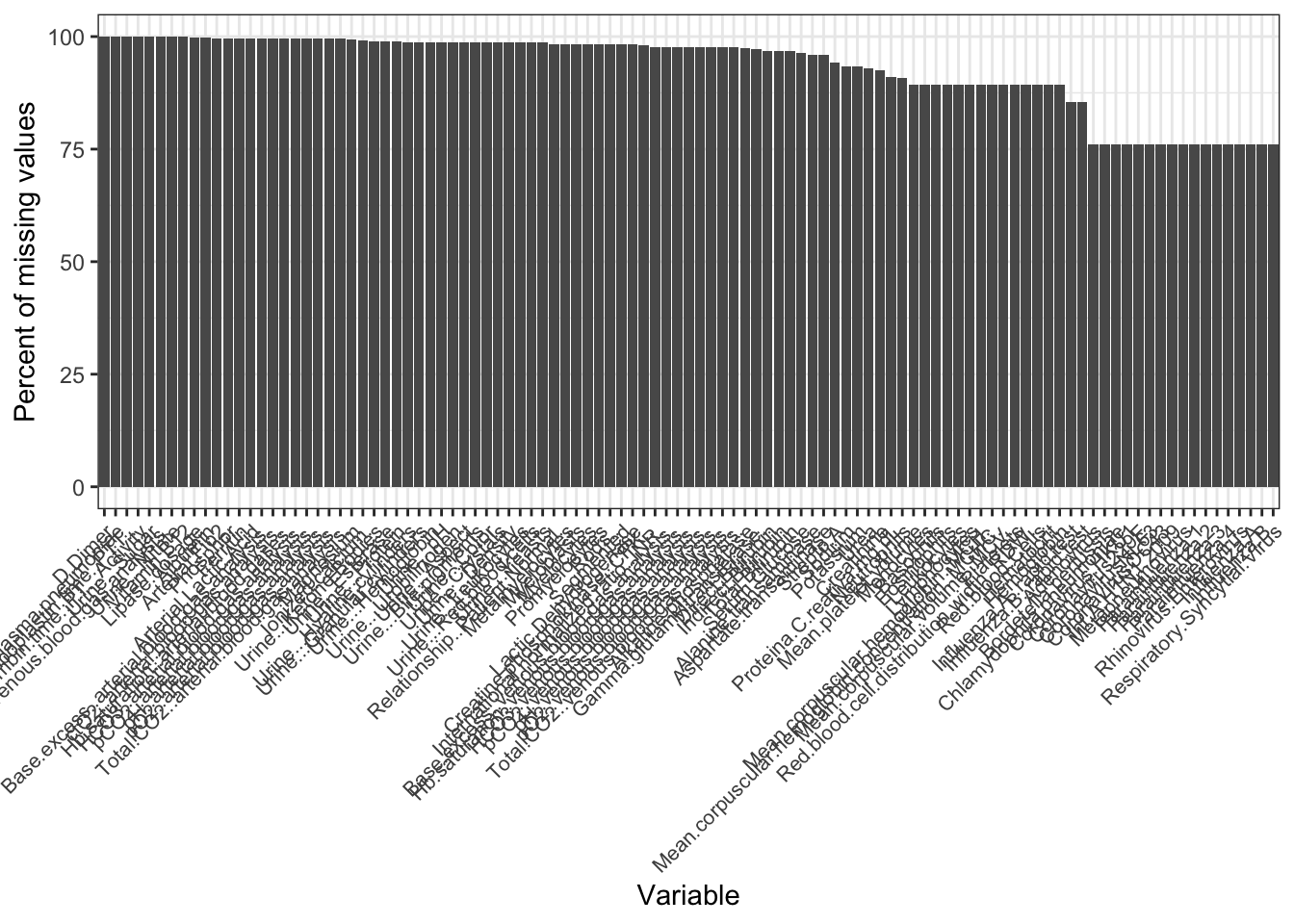
Este gráfico mostra quais são as variáveis com maior quantidade de dados faltantes. Note que algumas delas possuem quase 100% de dados faltantes! Veja o gráfico a seguir para que tenhamos uma ideia melhor da magnitude da falta de dados que estamos enfrentando:
vis_miss(dataset_clean) +
theme(axis.text.x = element_text(size = 6))

Mais de 91% de dados faltantes é muita coisa. Este tipo de informação não me dá muita esperança em encontrar um modelo bom para estes dados. Mesmo assim, vou tentar algo e ver o que acontece. Para isso, vou manter no conjunto a ser analisado apenas as colunas com pelo menos 1000 observações. Ou seja, vou retirar todas as colunas com muito poucos dados registrados.
# keep only columns with at least n observations
n <- 1000
dataset_clean <- dataset_clean[, which(dim(dataset_clean)[1] - apply(apply(dataset_clean, 2, is.na), 2, sum) >= n)]
# remove quantitative variables with variance equal to zero
dataset_model_num <- dataset_clean %>%
select_if(is.numeric)
if (sum(apply(dataset_model_num, 2, var, na.rm = TRUE) == 0) != 0) {
dataset_model_num <- dataset_model_num[, which(apply(dataset_model_num, 2, var, na.rm = TRUE) == 0)]
}
# remove categorical variables with only one level
dataset_model_cat <- dataset_clean %>%
select_if(negate(is.numeric))
dataset_model_cat <- dataset_model_cat[, sapply(dataset_model_cat, nlevels) > 1]
# final dataset
dataset_model <- base::cbind(dataset_model_num, dataset_model_cat)
vis_miss(dataset_model) + # it needs naniar package
theme(axis.text.x = element_text(size = 6))

É possíver ver que há uma proporção menor de dados faltantes neste conjunto processado. Baixamos a proporção de dados faltantes de 91,4% para 68%. Ainda é um valor bastante alto, mas está um pouco menos pior do que o original.
Abaixo coloco mais alguns gráficos que tentam relacionar as variáveis que restaram no conjunto de dados, mas aparentemente não há relação alguma entre elas.
# some other plots
ggpairs(dataset_model[, c(1:10)])
## `stat_bin()` using `bins = 30`. Pick better value with `binwidth`.
## `stat_bin()` using `bins = 30`. Pick better value with `binwidth`.
## Warning: Removed 4290 rows containing non-finite outside the scale range
## (`stat_g_gally_count()`).
## Removed 4290 rows containing non-finite outside the scale range
## (`stat_g_gally_count()`).
## Removed 4290 rows containing non-finite outside the scale range
## (`stat_g_gally_count()`).
## Removed 4290 rows containing non-finite outside the scale range
## (`stat_g_gally_count()`).
## Removed 4290 rows containing non-finite outside the scale range
## (`stat_g_gally_count()`).
## Removed 4290 rows containing non-finite outside the scale range
## (`stat_g_gally_count()`).
## Removed 4290 rows containing non-finite outside the scale range
## (`stat_g_gally_count()`).
## `stat_bin()` using `bins = 30`. Pick better value with `binwidth`.
## Warning: Removed 4290 rows containing non-finite outside the scale range
## (`stat_g_gally_count()`).
## Removed 4290 rows containing non-finite outside the scale range
## (`stat_g_gally_count()`).
## Removed 4290 rows containing non-finite outside the scale range
## (`stat_g_gally_count()`).
## Removed 4290 rows containing non-finite outside the scale range
## (`stat_g_gally_count()`).
## Removed 4290 rows containing non-finite outside the scale range
## (`stat_g_gally_count()`).
## Removed 4290 rows containing non-finite outside the scale range
## (`stat_g_gally_count()`).
## `stat_bin()` using `bins = 30`. Pick better value with `binwidth`.
## Warning: Removed 4290 rows containing non-finite outside the scale range
## (`stat_g_gally_count()`).
## Removed 4290 rows containing non-finite outside the scale range
## (`stat_g_gally_count()`).
## Removed 4290 rows containing non-finite outside the scale range
## (`stat_g_gally_count()`).
## Removed 4290 rows containing non-finite outside the scale range
## (`stat_g_gally_count()`).
## Removed 4290 rows containing non-finite outside the scale range
## (`stat_g_gally_count()`).
## `stat_bin()` using `bins = 30`. Pick better value with `binwidth`.
## Warning: Removed 4292 rows containing non-finite outside the scale range
## (`stat_g_gally_count()`).
## Warning: Removed 4292 rows containing non-finite outside the scale range
## (`stat_g_gally_count()`).
## Removed 4292 rows containing non-finite outside the scale range
## (`stat_g_gally_count()`).
## Removed 4292 rows containing non-finite outside the scale range
## (`stat_g_gally_count()`).
## `stat_bin()` using `bins = 30`. Pick better value with `binwidth`.
## Warning: Removed 4292 rows containing non-finite outside the scale range
## (`stat_g_gally_count()`).
## Removed 4292 rows containing non-finite outside the scale range
## (`stat_g_gally_count()`).
## Removed 4292 rows containing non-finite outside the scale range
## (`stat_g_gally_count()`).
## `stat_bin()` using `bins = 30`. Pick better value with `binwidth`.
## Warning: Removed 4292 rows containing non-finite outside the scale range
## (`stat_g_gally_count()`).
## Removed 4292 rows containing non-finite outside the scale range
## (`stat_g_gally_count()`).
## `stat_bin()` using `bins = 30`. Pick better value with `binwidth`.
## Warning: Removed 4292 rows containing non-finite outside the scale range
## (`stat_g_gally_count()`).
## `stat_bin()` using `bins = 30`. Pick better value with `binwidth`.

ggpairs(dataset_model[, c(11:ncol(dataset_model))])
## Warning: Removed 4292 rows containing non-finite outside the scale range
## (`stat_g_gally_count()`).
## Removed 4292 rows containing non-finite outside the scale range
## (`stat_g_gally_count()`).
## Removed 4292 rows containing non-finite outside the scale range
## (`stat_g_gally_count()`).
## Removed 4292 rows containing non-finite outside the scale range
## (`stat_g_gally_count()`).
## Removed 4292 rows containing non-finite outside the scale range
## (`stat_g_gally_count()`).
## Removed 4292 rows containing non-finite outside the scale range
## (`stat_g_gally_count()`).
## Removed 4292 rows containing non-finite outside the scale range
## (`stat_g_gally_count()`).
## Removed 4292 rows containing non-finite outside the scale range
## (`stat_g_gally_count()`).
## Removed 4292 rows containing non-finite outside the scale range
## (`stat_g_gally_count()`).
## Removed 4292 rows containing non-finite outside the scale range
## (`stat_g_gally_count()`).
## Removed 4292 rows containing non-finite outside the scale range
## (`stat_g_gally_count()`).
## Removed 4292 rows containing non-finite outside the scale range
## (`stat_g_gally_count()`).
## Removed 4292 rows containing non-finite outside the scale range
## (`stat_g_gally_count()`).
## Removed 4292 rows containing non-finite outside the scale range
## (`stat_g_gally_count()`).
## Removed 4292 rows containing non-finite outside the scale range
## (`stat_g_gally_count()`).
## Removed 4292 rows containing non-finite outside the scale range
## (`stat_g_gally_count()`).
## Removed 4292 rows containing non-finite outside the scale range
## (`stat_g_gally_count()`).
## Removed 4292 rows containing non-finite outside the scale range
## (`stat_g_gally_count()`).
## Removed 4292 rows containing non-finite outside the scale range
## (`stat_g_gally_count()`).
## Removed 4292 rows containing non-finite outside the scale range
## (`stat_g_gally_count()`).
## Removed 4292 rows containing non-finite outside the scale range
## (`stat_g_gally_count()`).
## Removed 4292 rows containing non-finite outside the scale range
## (`stat_g_gally_count()`).
## Removed 4292 rows containing non-finite outside the scale range
## (`stat_g_gally_count()`).
## Removed 4292 rows containing non-finite outside the scale range
## (`stat_g_gally_count()`).
## Removed 4292 rows containing non-finite outside the scale range
## (`stat_g_gally_count()`).
## Removed 4292 rows containing non-finite outside the scale range
## (`stat_g_gally_count()`).
## Removed 4292 rows containing non-finite outside the scale range
## (`stat_g_gally_count()`).
## Removed 4292 rows containing non-finite outside the scale range
## (`stat_g_gally_count()`).
# it seems there is no relation between any pair of variables
Modelagem 1 Link para o cabeçalho
Poucos modelos de aprendizagem de máquina conseguem lidar com dados faltantes. Por este motivo, decidi escolher um modelo chamado CART (Classification and Regression Tree - Árvore de Classificação e Regressão). Abaixo estão os resultados obtidos, após a separação do conjunto de dados original em treinamento e teste. Os resultados reportados foram obtidos após a aplicação do modelo ajustado no conjunto de teste. Para saber mais porque fazer isso, recomendo novamente meu post Tutorial: Como Fazer o Seu Primeiro Projeto de Data Science.
################
### modeling ###
################
# train/test split
covid <- dataset_model
set.seed(1)
index <- createDataPartition(covid$SARS.Cov.2.exam.result,
p = 0.75,
list = FALSE)
covid_train <- covid[ index, ]
covid_test <- covid[-index, ]
dim(covid_train)
table(covid_train$SARS.Cov.2.exam.result)
dim(covid_test)
table(covid_test$SARS.Cov.2.exam.result)
# parameters for cart
fitControl <- trainControl(method = "cv",
number = 5,
savePred = TRUE,
classProb = TRUE)
tune.grid <- expand.grid(mincriterion = seq(from = 0.01,
to = .99,
by = 0.01))
set.seed(1)
x <- covid_train %>%
select(-SARS.Cov.2.exam.result)
y <- covid_train %>%
select(SARS.Cov.2.exam.result) %>%
unlist()
covid_ctree <- train(x, y,
method = "ctree",
tuneGrid = tune.grid,
trControl = fitControl)
ggplot(covid_ctree)
prediction <- predict(covid_ctree, covid_test)
confusionMatrix(prediction, covid_test$SARS.Cov.2.exam.result)
## Confusion Matrix and Statistics
##
## Reference
## Prediction negative positive
## negative 1271 139
## positive 0 0
##
## Accuracy : 0.9014
## 95% CI : (0.8847, 0.9165)
## No Information Rate : 0.9014
## P-Value [Acc > NIR] : 0.5226
##
## Kappa : 0
##
## Mcnemar's Test P-Value : <2e-16
##
## Sensitivity : 1.0000
## Specificity : 0.0000
## Pos Pred Value : 0.9014
## Neg Pred Value : NaN
## Prevalence : 0.9014
## Detection Rate : 0.9014
## Detection Prevalence : 1.0000
## Balanced Accuracy : 0.5000
##
## 'Positive' Class : negative
##
# 90% accuracy seems good, but No Information Rate is also 90%
# so, using this model or a random process gives the same answer
# high sensitivity, but very low specificity :(
Note que obtive 90% de acurácia no meu modelo. Ou seja, ele acerta 90% das tentativas de classificar um paciente como positivo ou negativo em relação ao coronavírus. Parece um resultado muito bom, mas não é. Ele é péssimo, na realidade.
No conjunto de dados original, aproximadamente 90% dos pacientes não possuem coronavírus, enquanto 10% estão infectados. O meu modelo encontra, com 100% de certeza, quem não tem coronavírus (é o valor de Sensitivity no output acima). Entretanto, encontra 0% dos pacientes com coronavírus (é o valor de Specificity no output acima). Ou seja, ele vai mandar para casa todo mundo que chegar no hospital, tendo coronavírus ou não.
Isso me fez partir para uma segunda modelagem.
Modelagem 2 Link para o cabeçalho
Vimos que a primeira modelagem não deu bons resultados. Com o intuito de tentar obter resultados melhores, vou proceder com a imputação de dados. Se for tentar rodar o código abaixo em seu computador, prepare-se para esperar uns bons minutos.
#######################
### data imputation ###
#######################
covid_imp <- mice(covid, meth = "rf", ntree = 5) # be patient
covid <- complete(covid_imp)
################
### modeling ###
################
# train/test split
set.seed(1)
index <- createDataPartition(covid$SARS.Cov.2.exam.result,
p = 0.75,
list = FALSE)
covid_train <- covid[ index, ]
covid_test <- covid[-index, ]
dim(covid_train)
table(covid_train$SARS.Cov.2.exam.result)
dim(covid_test)
table(covid_test$SARS.Cov.2.exam.result)
# parameters for random forest
fitControl <- trainControl(method = "cv",
number = 5,
savePred = TRUE,
classProb = TRUE)
tune.grid <- expand.grid(mtry = 1:35)
set.seed(1)
x <- covid_train %>%
select(-SARS.Cov.2.exam.result)
y <- covid_train %>%
select(SARS.Cov.2.exam.result) %>%
unlist()
covid_rf <- train(x, y,
method = "rf",
tuneGrid = tune.grid,
trControl = fitControl)
ggplot(covid_rf)
prediction <- predict(covid_rf, covid_test)
confusionMatrix(prediction, covid_test$SARS.Cov.2.exam.result)
## Confusion Matrix and Statistics
##
## Reference
## Prediction negative positive
## negative 1271 139
## positive 0 0
##
## Accuracy : 0.9014
## 95% CI : (0.8847, 0.9165)
## No Information Rate : 0.9014
## P-Value [Acc > NIR] : 0.5226
##
## Kappa : 0
##
## Mcnemar's Test P-Value : <2e-16
##
## Sensitivity : 1.0000
## Specificity : 0.0000
## Pos Pred Value : 0.9014
## Neg Pred Value : NaN
## Prevalence : 0.9014
## Detection Rate : 0.9014
## Detection Prevalence : 1.0000
## Balanced Accuracy : 0.5000
##
## 'Positive' Class : negative
##
# high sensitivity, but very low specificity :(
De novo, o mesmo problema: alta sensitividade e baixíssima especificidade neste modelo. Ou seja, não é melhor do que o modelo anterior ou uma seleção aleatória de diagnóstico.
Conclusão Link para o cabeçalho
Eu cheguei no meu modelo final. 90% de acurácia usando random forest. Mas como a variável resposta é desbalanceada, com 90% para uma classe e 10% para outra, este meu modelo não serve pra nada.
Mesmo tentando abordagens diversas, como filtragem de dados faltantes e imputação, nada deu certo pra mim. Aparentemente este é um resultado geral para este problema. Os outros participantes deste desafio no kaggle criaram várias análises diferentes, com muitas abordagens interessantes, mas ninguém consegue uma boa detecção de verdadeiros positivos.
Tenho pouca esperança que, com este conjunto de dados específico, seja possível fazer algo preditivo de qualidade. Talvez com uma feature engineering muito boa? Pode ser, mas não podemos esquecer que são muitos dados faltantes. No conjunto original, sem pré-processamento, são 91% de dados faltantes. Não dá pra fazer milagre.
Como sempre, o código utilizado nesta análise está disponível em meu github.