Quem nunca precisou criar um histograma de grupos diferentes no ggplot2 e se deparou com um resultado assim?
library(palmerpenguins)
library(tidyverse)
## ── Attaching core tidyverse packages ──────────────────────── tidyverse 2.0.0 ──
## ✔ dplyr 1.1.4 ✔ readr 2.1.5
## ✔ forcats 1.0.0 ✔ stringr 1.5.1
## ✔ ggplot2 3.5.1 ✔ tibble 3.2.1
## ✔ lubridate 1.9.3 ✔ tidyr 1.3.1
## ✔ purrr 1.0.2
## ── Conflicts ────────────────────────────────────────── tidyverse_conflicts() ──
## ✖ dplyr::filter() masks stats::filter()
## ✖ dplyr::lag() masks stats::lag()
## ℹ Use the conflicted package (<http://conflicted.r-lib.org/>) to force all conflicts to become errors
theme_set(theme_minimal())
ggplot(penguins, aes(x = bill_length_mm, fill = species)) +
geom_histogram() +
scale_fill_viridis_d()
## `stat_bin()` using `bins = 30`. Pick better value with `binwidth`.
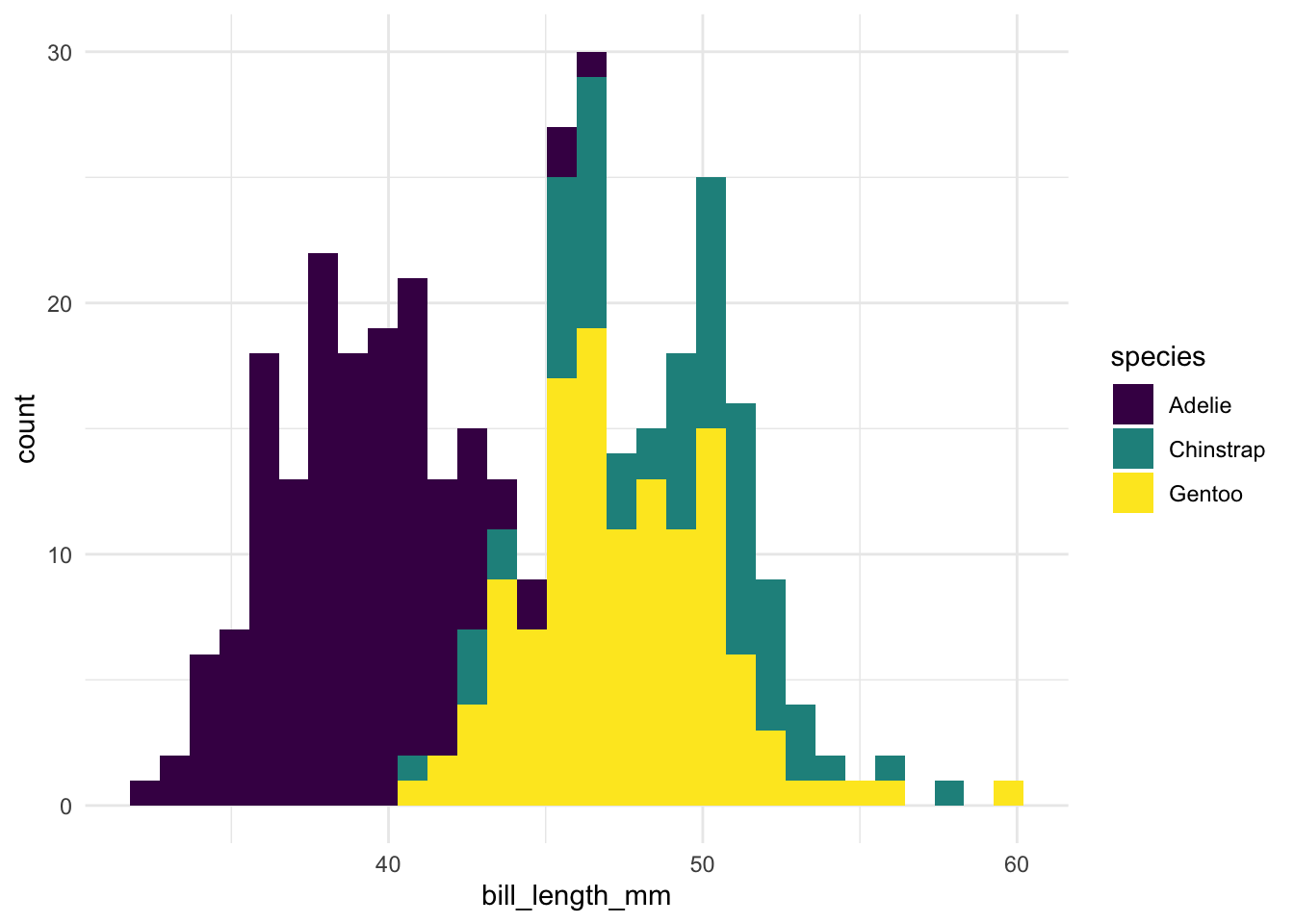
Note como os gráficos ficam empilhados, em vez de sobrepostos. Na minha opinião, isso prejudica muito a visualização dos dados e da sobreposição dos grupos, principalmente quando dois ou mais estão presentes.
Uma forma de evitar isso é adicionando uma transparência às colunas e alterando o argumento position para identity:
ggplot(penguins, aes(x = bill_length_mm, fill = species)) +
geom_histogram(alpha = 0.5, position = "identity") +
scale_fill_viridis_d()
## `stat_bin()` using `bins = 30`. Pick better value with `binwidth`.
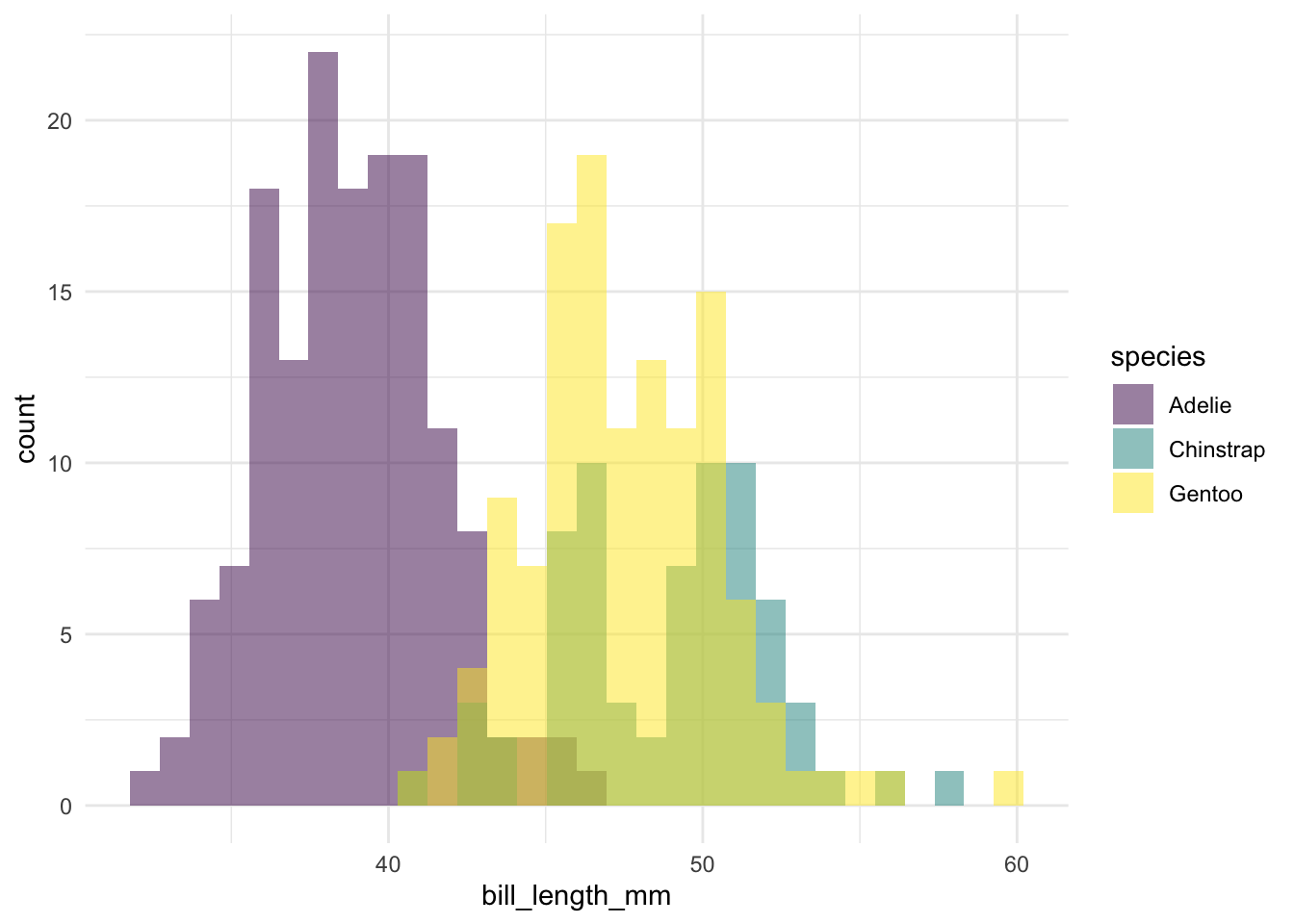
Perceba como agora é possível diferenciar bem entre os grupos e verificar facilmente onde eles estão se sobrepondo. Para mim, é a melhor maneira de utilizar este tipo de recurso.
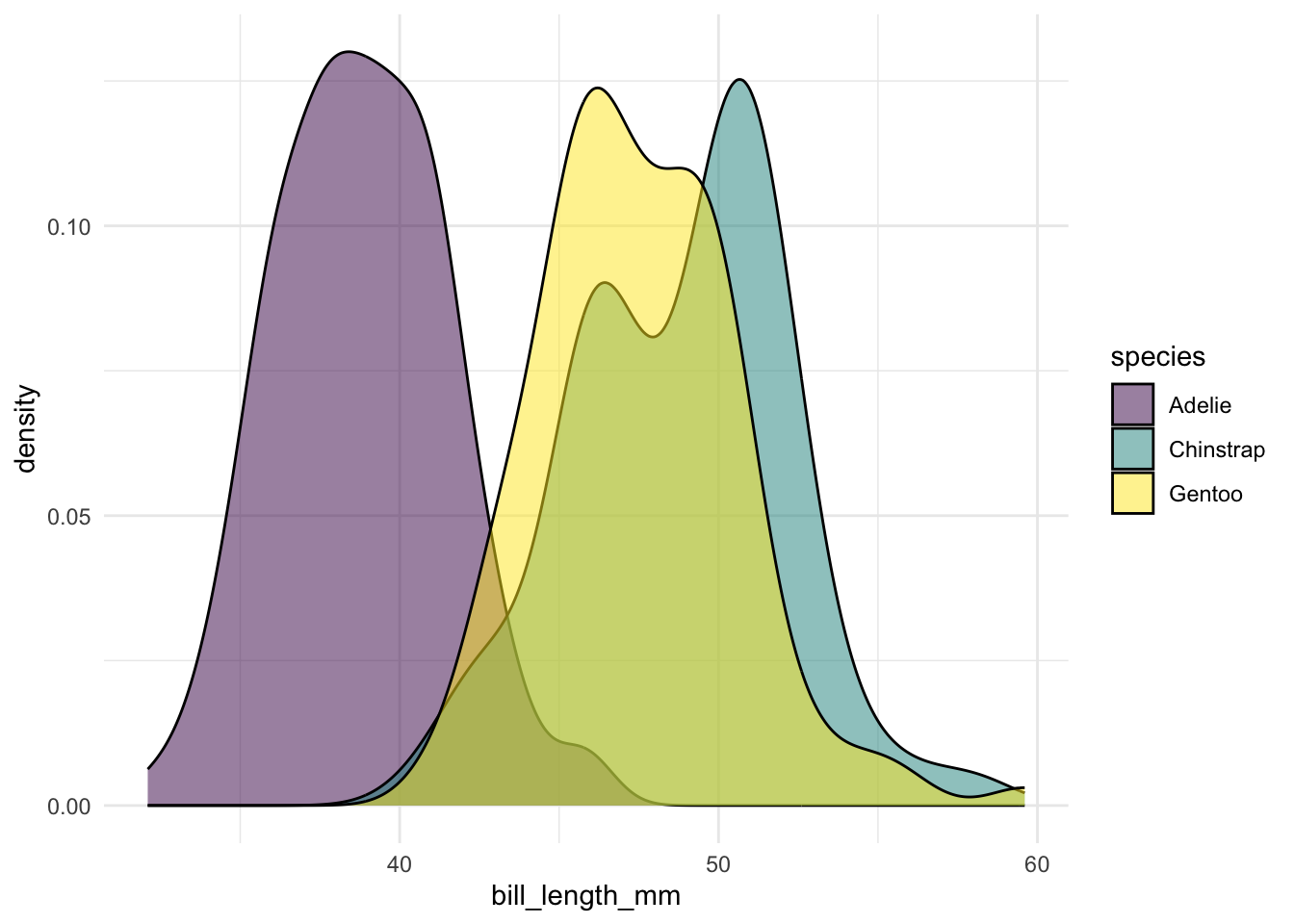
Mas, na verdade, prefiro ainda mais utilizar geom_density. Acho que o resultado é ainda melhor do que aquele obtido com geom_density:
ggplot(penguins, aes(x = bill_length_mm, fill = species)) +
geom_density(alpha = 0.5) +
scale_fill_viridis_d()