Uma forma de inclusão para pessoas com algum tipo de distúrbio da visão é utilizar paletas de cores inclusivas. Uma dessas paletas é chamada viridis, que é a minha paleta de cores preferida para criar a maior parte das minhas visualização de dados.
Nesse post usarei o pacote colorspace para simular diferentes três tipos de daltonismo: protanomalia (deficiência de visão vermelha), deuteranomalia (deficiência de visão verde) e tritanomalia (deficiência de visao azul). Com a simulação feita, irei comparar casa deficiência em seu grau mais severo com a paleta de cores padrão do ggplot2 e a paleta de cores viridis.
Preparação Link para o cabeçalho
A primeira parte da análise consiste em uma preparação básica: caregar pacotes e criar as paletas de cores original do ggplot2 e viridis.
library(colorspace)
library(tidyverse)
theme_set(theme_bw())
library(palmerpenguins)
library(patchwork)
# construcao da paleta padrao
padrao <-
ggplot(penguins, aes(x = flipper_length_mm, y = body_mass_g, colour = species)) +
geom_point() +
labs(x = "Comprimento da Nadadeira (mm)", y = "Massa Corporal (g)", colour = "Espécie", title = "Paleta de cores padrão")
ggplot_build(padrao)$data |>
first() |>
select(colour) |>
distinct()
## colour
## 1 #F8766D
## 2 #619CFF
## 3 #00BA38
paleta_padrao <- c("#F8766D", "#619CFF", "#00BA38")
# construcao da paleta viridis
viridis <-
ggplot(penguins, aes(x = flipper_length_mm, y = body_mass_g, colour = species)) +
geom_point() +
labs(x = "Comprimento da Nadadeira (mm)", y = "Massa Corporal (g)", colour = "Espécie", title = "Paleta de cores viridis") +
scale_colour_viridis_d()
ggplot_build(viridis)$data |>
first() |>
select(colour) |>
distinct()
## colour
## 1 #440154FF
## 2 #FDE725FF
## 3 #21908CFF
paleta_viridis <- c("#440154", "#FDE725", "#21908C")
Com isso pronto, basta gerar as paletas com as cores
Protanomalia: Deficiência de visão vermelha Link para o cabeçalho
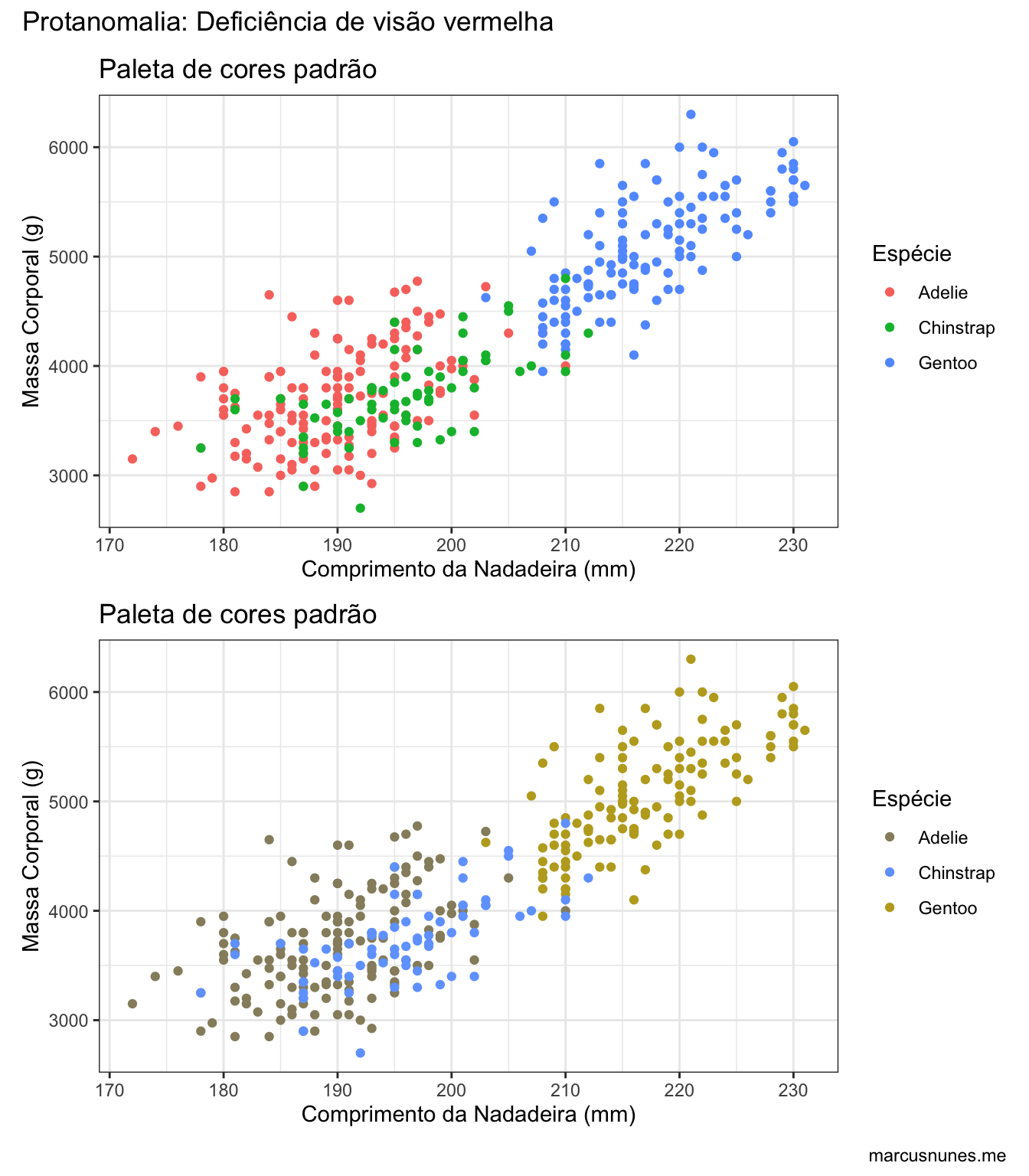
paleta_padrao_protanomalia <- simulate_cvd(paleta_padrao, protanomaly_cvd[11][[1]])
padrao + padrao +
scale_colour_manual(values = paleta_padrao_protanomalia) +
plot_layout(nrow = 2) +
plot_annotation(title = "Protanomalia: Deficiência de visão vermelha", caption = "marcusnunes.me")
paleta_viridis_protanomalia <- simulate_cvd(paleta_viridis, protanomaly_cvd[11][[1]])
viridis + viridis +
scale_colour_manual(values = paleta_viridis_protanomalia) +
plot_layout(nrow = 2) +
plot_annotation(title = "Protanomalia: Deficiência de visão vermelha", caption = "marcusnunes.me")
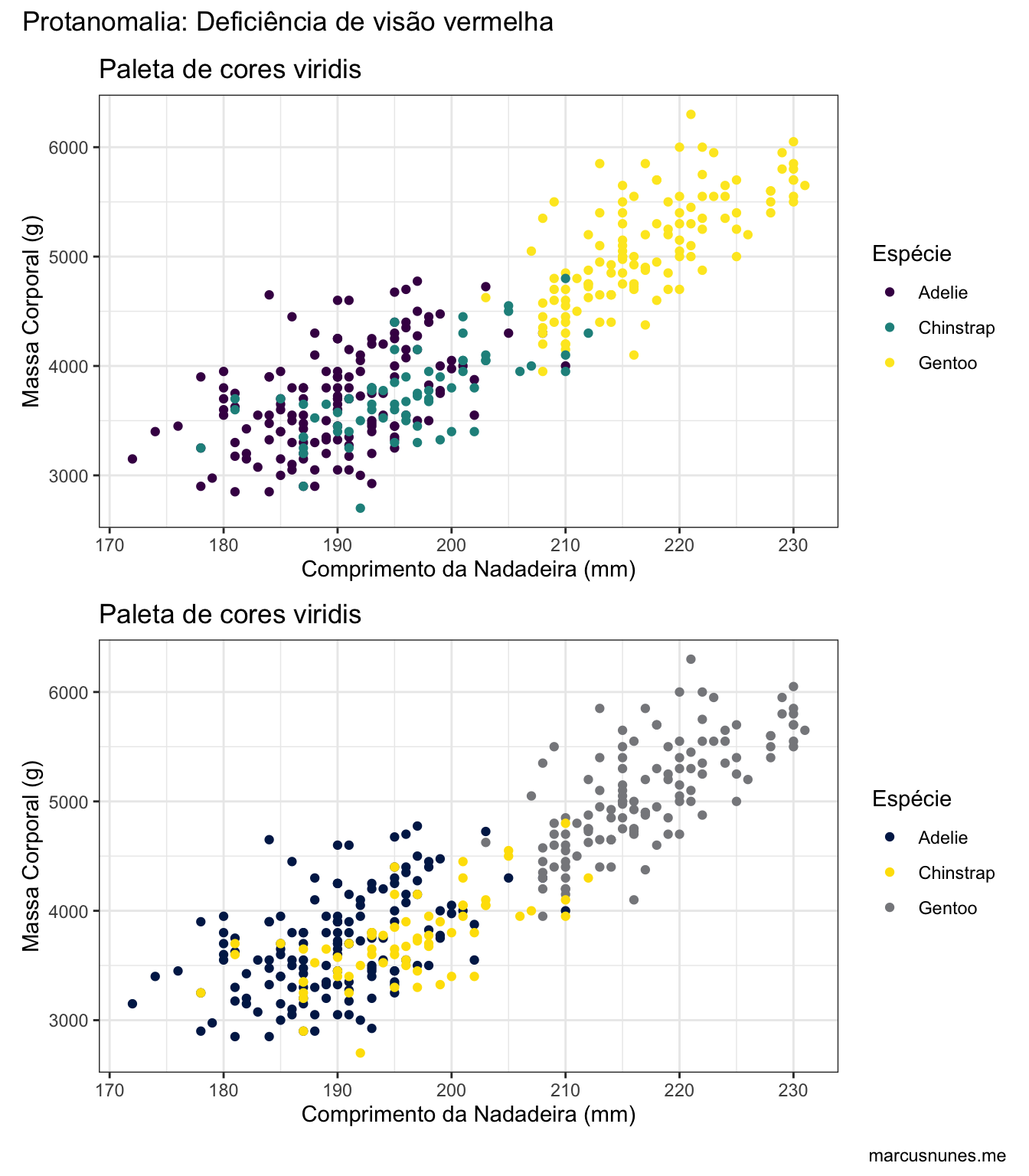
Note como na paleta original as espécies Adeli e Gentoo se confundem.
Deuteranomalia - Deficiência de visão verde Link para o cabeçalho
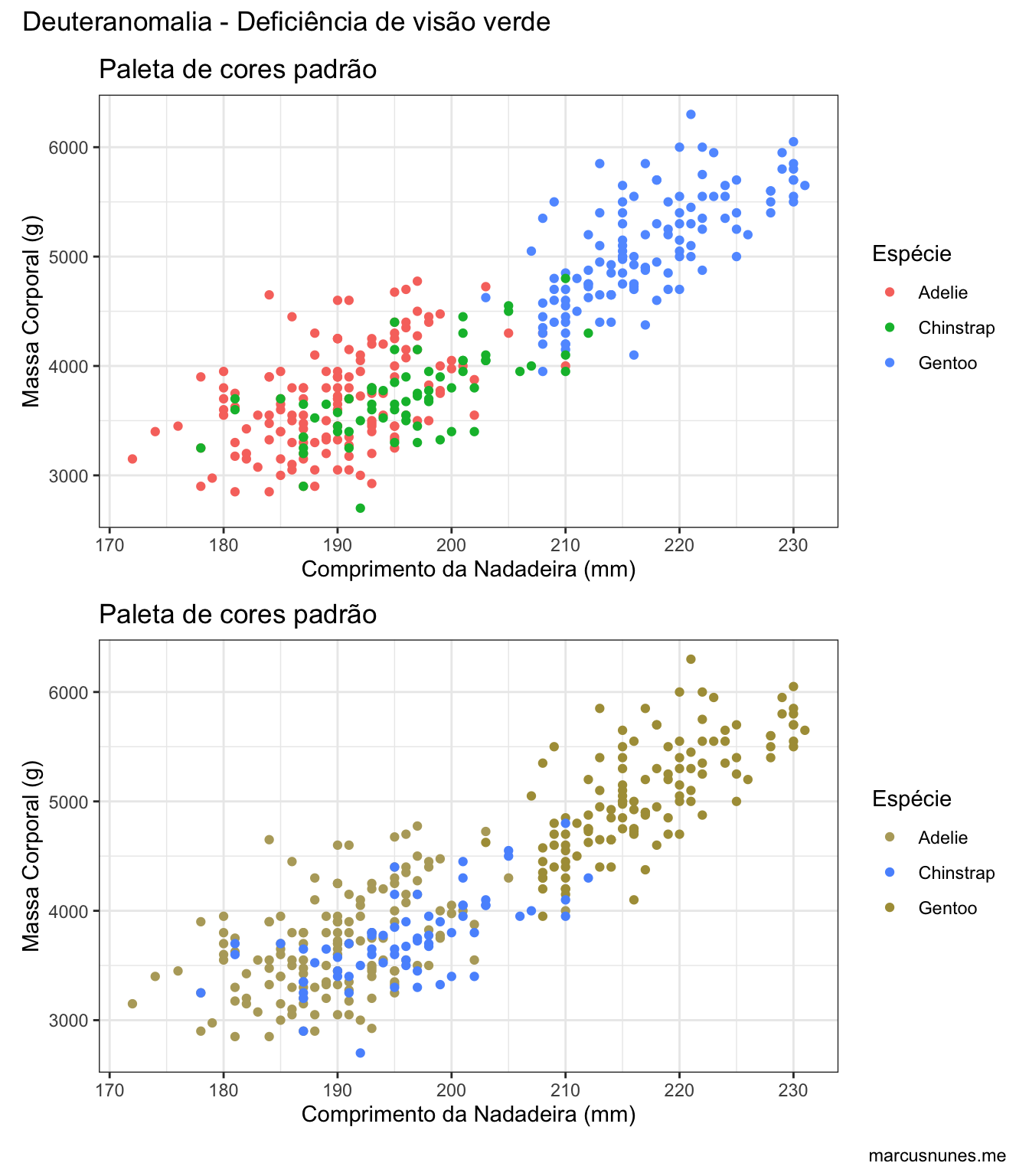
paleta_padrao_deuteranomalia <- simulate_cvd(paleta_padrao, deutanomaly_cvd[11][[1]])
padrao + padrao +
scale_colour_manual(values = paleta_padrao_deuteranomalia) +
plot_layout(nrow = 2) +
plot_annotation(title = "Deuteranomalia - Deficiência de visão verde", caption = "marcusnunes.me")
paleta_viridis_deuteranomalia <- simulate_cvd(paleta_viridis, deutanomaly_cvd[11][[1]])
viridis + viridis +
scale_colour_manual(values = paleta_viridis_deuteranomalia) +
plot_layout(nrow = 2) +
plot_annotation(title = "Deuteranomalia - Deficiência de visão verde", caption = "marcusnunes.me")
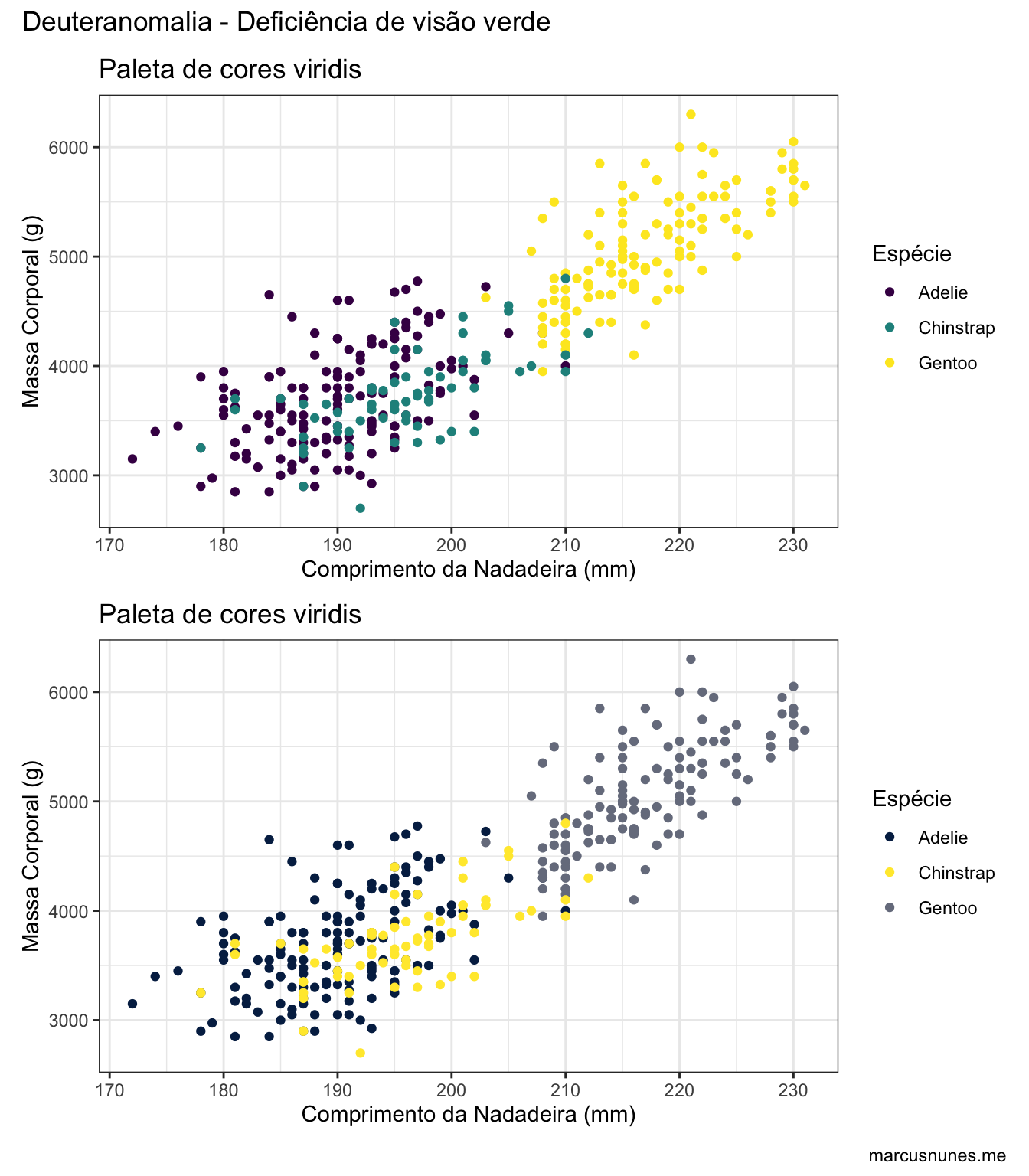
Note como na paleta original, novamente as espécies Adeli e Gentoo se confundem.
Tritanomalia - Deficiência de visão azul Link para o cabeçalho
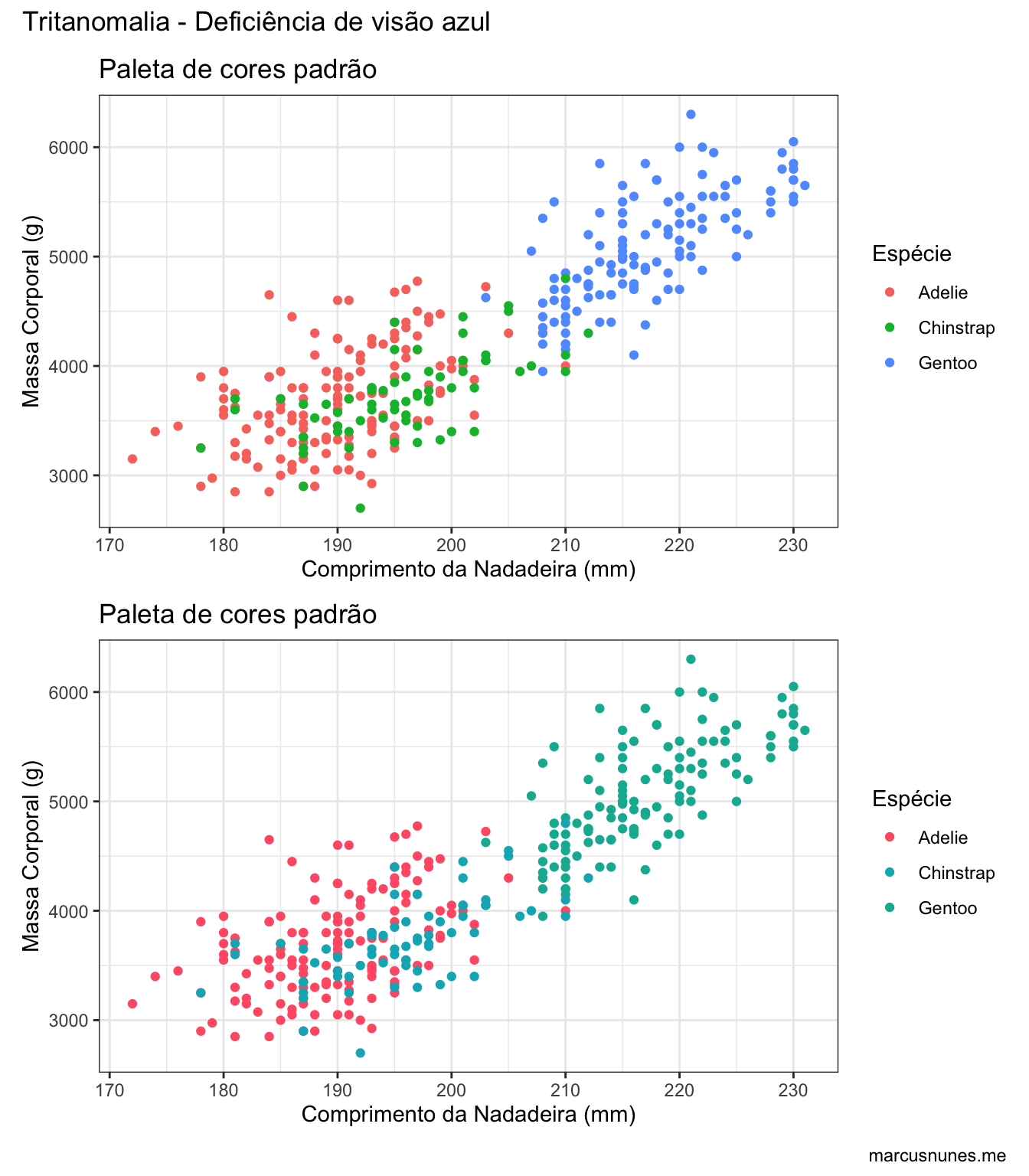
paleta_padrao_tritanomalia <- simulate_cvd(paleta_padrao, tritanomaly_cvd[11][[1]])
padrao + padrao +
scale_colour_manual(values = paleta_padrao_tritanomalia) +
plot_layout(nrow = 2) +
plot_annotation(title = "Tritanomalia - Deficiência de visão azul", caption = "marcusnunes.me")
paleta_viridis_tritanomalia <- simulate_cvd(paleta_viridis, tritanomaly_cvd[11][[1]])
viridis + viridis +
scale_colour_manual(values = paleta_viridis_tritanomalia) +
plot_layout(nrow = 2) +
plot_annotation(title = "Tritanomalia - Deficiência de visão azul", caption = "marcusnunes.me")
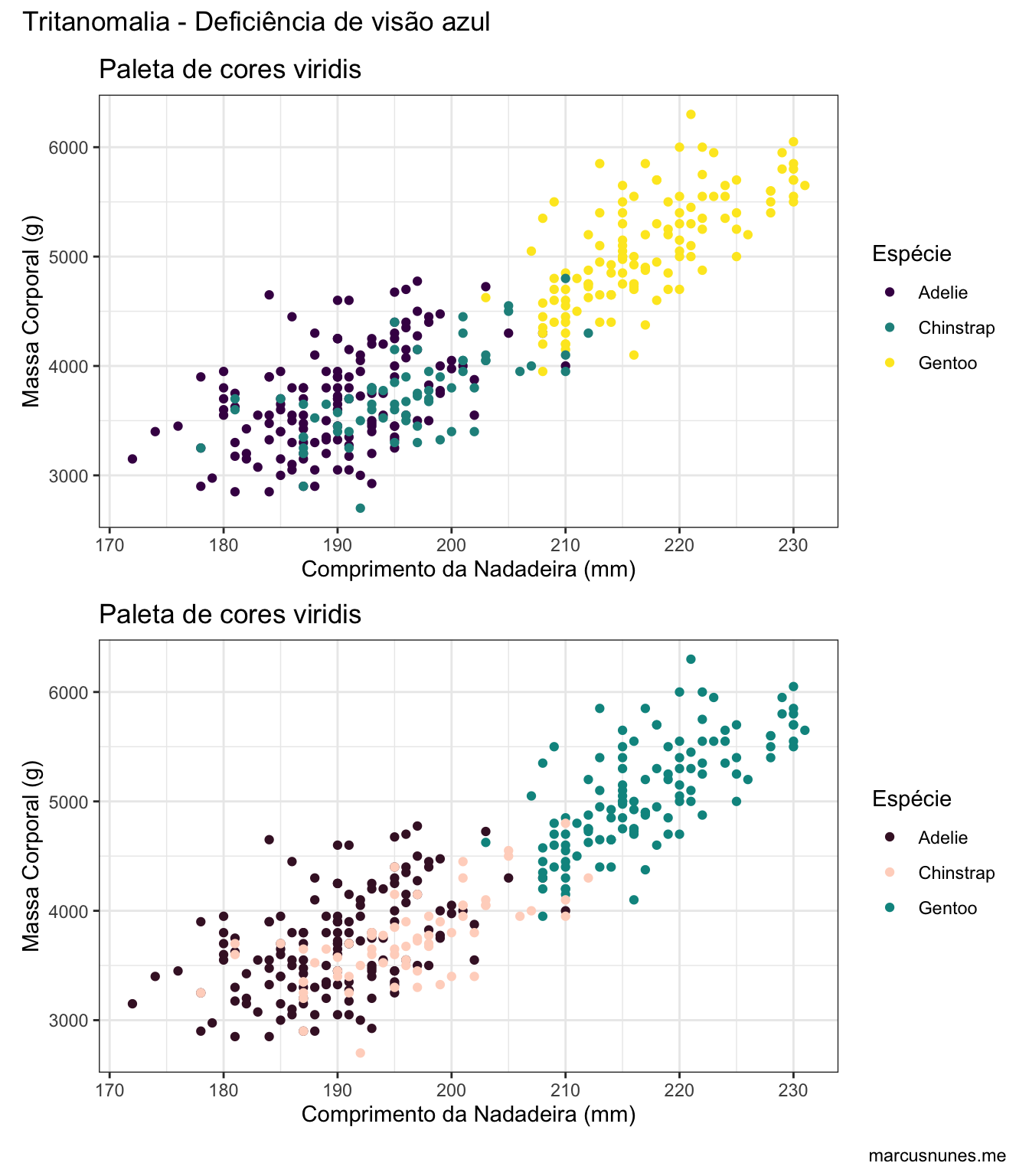
Note como na paleta original, mais uma vez as espécies Adeli e Gentoo se confundem.
Post baseado no paper colorspace: A Toolbox for Manipulating and Assessing Colors and Palettes.