Introdução Link para o cabeçalho
Decidi usar novamente dados do SUS para criar uma nova versão de um dos posts de maior sucesso deste blog chamado Heatmap: Os Aniversários Mais Comuns no Brasil . Entretanto, dessa vez, vou calcular os dias com maior ocorrências de óbitos no país entre 1996 e 2023.
Análise Link para o cabeçalho
Os dados são baixados através do pacote microdatasus. Quem estiver fora do Brasil não poderá usar o pacote e deverá baixar os arquivos no S3 DataSUS FTP mirror, site mantido pelo autor do pacote.
# pacotes necessarios
library(tidyverse)
library(janitor)
#devtools::install_github("rfsaldanha/microdatasus")
library(microdatasus)
Depois dos pacotes carreagados, é necessário determinar quais UF terão seus dados baixados.
uf <- c("AC", "AL", "AP", "AM", "BA", "CE", "DF", "ES", "GO",
"MA", "MT", "MS", "MG", "PA", "PB", "PR", "PE", "PI",
"RJ", "RN", "RS", "RO", "RR", "SC", "SP", "SE", "TO")
A função fetch_datasus vai baixar os dados de cada UF, para os anos determinados na chamada da função. Para economizar banda e espaço em disco, vou baixar apenas as variáveis sobre data de óbito (DTOBITO) e data de nascimento (DTNASC).
for (j in uf){
dados <- fetch_datasus(year_start = 1996,
year_end = 2023,
uf = j,
information_system = "SIM-DO",
vars = c("DTOBITO", "DTNASC"))
saveRDS(object = dados, file = paste(j, ".rds", sep = ""))
}
Com os dados baixados, é necessário lê-los no R. Para isso, criei um loop e salvei tudo em um arquivo chamado obitos.RData:
# preparacao dos dados
arquivos <- list.files("dados")
arquivos <- grep(".dbc", arquivos, value = TRUE)
library(read.dbc)
dados <- read.dbc(file = paste0("dados/", arquivos[1]))
nascimento <- as.character(dados$DTNASC)
obito <- as.character(dados$DTOBITO)
for (j in arquivos[-1]) {
dados <- read.dbc(file = paste0("dados/", j))
nascimento <- c(nascimento, as.character(dados$DTNASC))
obito <- c(obito, as.character(dados$DTOBITO))
print(j)
}
## [1] "DOBR1997.dbc"
## [1] "DOBR1998.dbc"
## [1] "DOBR1999.dbc"
## [1] "DOBR2000.dbc"
## [1] "DOBR2001.dbc"
## [1] "DOBR2002.dbc"
## [1] "DOBR2003.dbc"
## [1] "DOBR2004.dbc"
## [1] "DOBR2005.dbc"
## [1] "DOBR2006.dbc"
## [1] "DOBR2007.dbc"
## [1] "DOBR2008.dbc"
## [1] "DOBR2009.dbc"
## [1] "DOBR2010.dbc"
## [1] "DOBR2011.dbc"
## [1] "DOBR2012.dbc"
## [1] "DOBR2013.dbc"
## [1] "DOBR2014.dbc"
## [1] "DOBR2015.dbc"
## [1] "DOBR2016.dbc"
## [1] "DOBR2017.dbc"
## [1] "DOBR2018.dbc"
## [1] "DOBR2019.dbc"
## [1] "DOBR2020.dbc"
## [1] "DOBR2021.dbc"
## [1] "DOBR2022.dbc"
## [1] "DOBR2023.dbc"
dados <- data.frame(nascimento, obito)
save(dados, file = "dados/obitos.RData")
Gráfico Link para o cabeçalho
Para criar o heatmap de maneira que fosse informativo, pensei em calcular a média diária de nascimentos por dia. A partir disso, calculei a diferença percentual entre cada dia do ano e a média anual. Pintei com uma cor clara os valores maiores e com uma cor escura os menores. Além disso, colori de branco os valores sem diferença em relação à média, de modo que houvesse uma cor de referência.
Como considerei 29 de fevereiro na análise, fiz uma compensação proporcional para o resultado desse dia, considerando que no período de 28 anos analisado houve 7 anos bissextos.
# pacotes necessarios
library(tidyverse)
library(viridis)
# leitura dos dados
load("dados/obitos.RData")
dias <- substr(dados$obito, 1, 2)
meses <- substr(dados$obito, 3, 4)
datas <- substr(dados$obito, 1, 4)
dias_do_ano <-
c(sprintf("%04s", paste(01:31, "01", sep = "")),
sprintf("%04s", paste(01:31, "03", sep = "")),
sprintf("%04s", paste(01:31, "05", sep = "")),
sprintf("%04s", paste(01:31, "07", sep = "")),
sprintf("%04s", paste(01:31, "08", sep = "")),
sprintf("%04s", paste(01:31, "10", sep = "")),
sprintf("%04s", paste(01:31, "12", sep = "")),
sprintf("%04s", paste(01:30, "04", sep = "")),
sprintf("%04s", paste(01:30, "06", sep = "")),
sprintf("%04s", paste(01:30, "09", sep = "")),
sprintf("%04s", paste(01:30, "11", sep = "")),
sprintf("%04s", paste(01:29, "02", sep = "")))
datas <- datas[datas %in% dias_do_ano]
datas_tabela <- table(datas)
datas_tabela_proporcao <- as.vector(datas_tabela/mean(datas_tabela)-1)*100
summary(datas_tabela_proporcao)
## Min. 1st Qu. Median Mean 3rd Qu. Max.
## -76.9457 -3.2494 -0.8259 0.0000 3.6544 10.2971
# criacao da malha de datas
meses <- c("Janeiro", "Fevereiro", "Março", "Abril", "Maio", "Junho", "Julho", "Agosto", "Setembro", "Outubro", "Novembro", "Dezembro")
malha <- expand.grid(x = meses, y = 1:31)
malha <-
malha |>
arrange(y)
# retirar dias que nao existem
malha <-
malha |>
filter(!(x == "Fevereiro" & y > 29)) |>
filter(!(x %in% c("Abril", "Junho", "Setembro", "Novembro") & y > 30)) |>
mutate(x = factor(x,
levels = c("Janeiro", "Fevereiro", "Março", "Abril", "Maio", "Junho", "Julho", "Agosto", "Setembro", "Outubro", "Novembro", "Dezembro")))
# colocar as porcentagens dos nascimentos
malha <- data.frame(malha, prop = datas_tabela_proporcao)
# compensacao de 29 de fevereiro
malha[338, 3] <- malha[338, 3]*(7/28)
# grafico
ggplot(malha, aes(x = y, y = fct_rev(x), fill = prop)) +
geom_tile(colour = "grey10") +
scale_fill_gradient2(low = viridis(2)[1], high = viridis(2)[2], mid = "white", midpoint = 0) +
scale_x_continuous(breaks = 1:31) +
coord_equal() +
theme_minimal() +
theme(panel.grid.major = element_blank(), panel.grid.minor = element_blank()) +
labs(x = "", y = "", fill = "Diferença Percentual\nem Relação à Média", title = "As datas de óbito mais comuns no Brasil: 1996-2023", caption = "https://marcusnunes.me")
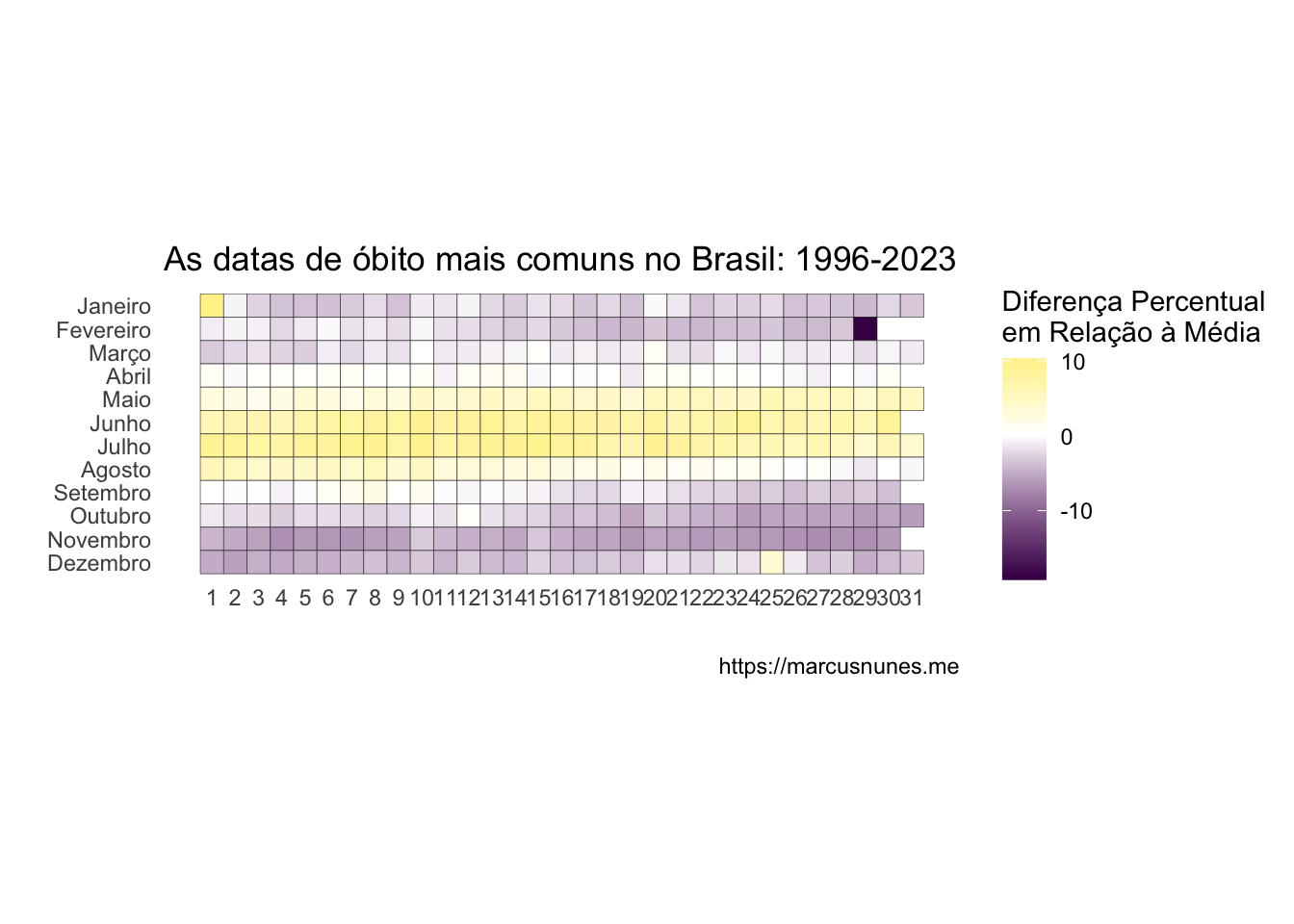
Inferências sobre a Imagem Link para o cabeçalho
Os pontos escuros espalhados no grid são os primeiros detalhes que me chamam a atenção. Ao checarmos quais são os 10 dias de óbitos menos comuns, temos o seguinte:
# dias menos comuns
malha |>
arrange(prop) |>
head(10) |>
mutate(prop = round(prop, 2))
## x y prop
## 1 Fevereiro 29 -19.24
## 2 Novembro 27 -6.99
## 3 Novembro 29 -6.89
## 4 Novembro 4 -6.76
## 5 Novembro 26 -6.67
## 6 Novembro 28 -6.52
## 7 Novembro 7 -6.39
## 8 Novembro 19 -6.35
## 9 Novembro 6 -6.32
## 10 Novembro 24 -6.18
Com exceção de 29 de fevereiro, todos os dias menos frequentes são em novembro. Ao contrário do que inferi sobre os nascimentos, não consegui estabelecer nenhuma hipótese sobre o porquê disso.
Os 10 dias mais comuns, por outro lado, têm a seguinte configuração:
# dias mais comuns
malha |>
arrange(desc(prop)) |>
head(10) |>
mutate(prop = round(prop, 2))
## x y prop
## 1 Janeiro 1 10.30
## 2 Julho 10 9.81
## 3 Julho 15 9.43
## 4 Julho 8 9.37
## 5 Julho 7 9.16
## 6 Junho 10 9.06
## 7 Julho 14 9.03
## 8 Julho 20 9.00
## 9 Julho 13 8.97
## 10 Julho 1 8.95
Todos ocorrem em julho, exceto pelo dia 1 de Janeiro. Me questiono se isso tem a ver com o Reveillón e excesso de consumo de bebidas e drogas.
Uma segunda versão desse heatmap poderia utilizar apenas dados de pessoas mortas devido a causas externas.