Vamos supor que eu desejo criar boxplots para cada variável resposta e cada espécie presente no conjunto de dados iris. Além disso, quero utilizar o ggplot para essa tarefa. A primeira tentativa a ser feita seria algo como o seguinte:
library(tidyverse)
## ── Attaching core tidyverse packages ──────────────────────── tidyverse 2.0.0 ──
## ✔ dplyr 1.1.4 ✔ readr 2.1.5
## ✔ forcats 1.0.0 ✔ stringr 1.5.1
## ✔ ggplot2 3.5.0 ✔ tibble 3.2.1
## ✔ lubridate 1.9.3 ✔ tidyr 1.3.1
## ✔ purrr 1.0.2
## ── Conflicts ────────────────────────────────────────── tidyverse_conflicts() ──
## ✖ dplyr::filter() masks stats::filter()
## ✖ dplyr::lag() masks stats::lag()
## ℹ Use the conflicted package (<http://conflicted.r-lib.org/>) to force all conflicts to become errors
theme_set(theme_bw())
library(patchwork)
g1 <-
ggplot(iris, aes(x = Species, y = Petal.Length)) +
geom_boxplot()
g2 <-
ggplot(iris, aes(x = Species, y = Petal.Width)) +
geom_boxplot()
g3 <-
ggplot(iris, aes(x = Species, y = Sepal.Length)) +
geom_boxplot()
g4 <-
ggplot(iris, aes(x = Species, y = Sepal.Width)) +
geom_boxplot()
g1 + g2 + g3 + g4
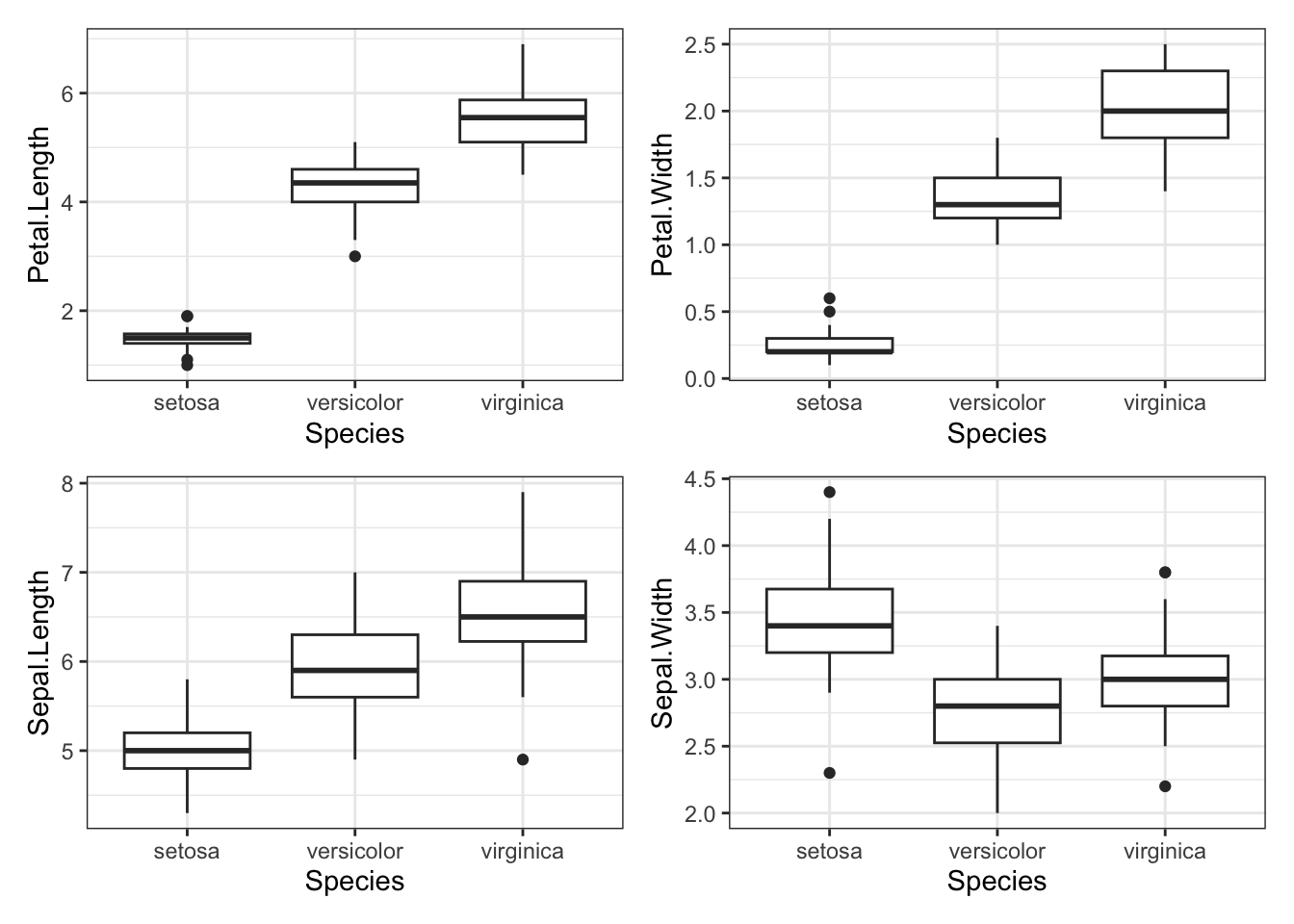
O problema dessa abordagem é o excesso de código escrito. Foi necessário criar quatro gráficos diferentes, um para cada variável, e juntá-los depois em uma figura apenas. Imagine o trabalho que seria se fossem mais do que quatro variáveis resposta.
Uma maneira mais fácil de resolver isso é converter dados do formato wide para long. A função pivot_longer, do pacote tidyr (já carregado através do tidyverse), serve justamente para isso.
Primeiro, vamos ver como é a estrutura do conjunto de dados iris, em suas seis primeiras linhas:
head(iris)
## Sepal.Length Sepal.Width Petal.Length Petal.Width Species
## 1 5.1 3.5 1.4 0.2 setosa
## 2 4.9 3.0 1.4 0.2 setosa
## 3 4.7 3.2 1.3 0.2 setosa
## 4 4.6 3.1 1.5 0.2 setosa
## 5 5.0 3.6 1.4 0.2 setosa
## 6 5.4 3.9 1.7 0.4 setosa
Queremos converter essas cinco colunas em três:
Species: permanece inalterada, indicando a espécie da planta;Variavel: nome da variável resposta de interesse;Valores: valor correspondente à espécie e à variável resposta.
Ou seja, algo assim:
## # A tibble: 6 × 3
## Species Variavel Valores
## <fct> <chr> <dbl>
## 1 setosa Sepal.Length 5.1
## 2 setosa Sepal.Width 3.5
## 3 setosa Petal.Length 1.4
## 4 setosa Petal.Width 0.2
## 5 setosa Sepal.Length 4.9
## 6 setosa Sepal.Width 3
Para obtermos esse resultado, basta utilizarmos a função pivot_longer. Precisamos informar os dados que queremos utilizar (iris), em quais colunas queremos aplicar a transformação (nesse caso, todas menos Species, ou seja, !Species) e em quais colunas colocaremos os nomes das variáveis resposta (names_to) e valores (values_to). O código, portanto, é o seguinte:
iris |>
pivot_longer(!Species,
names_to = "Variavel",
values_to = "Valores") |>
print(n = 6)
## # A tibble: 600 × 3
## Species Variavel Valores
## <fct> <chr> <dbl>
## 1 setosa Sepal.Length 5.1
## 2 setosa Sepal.Width 3.5
## 3 setosa Petal.Length 1.4
## 4 setosa Petal.Width 0.2
## 5 setosa Sepal.Length 4.9
## 6 setosa Sepal.Width 3
## # ℹ 594 more rows
Juntando isso com o gráfico, temos o seguinte resultado:
iris |>
pivot_longer(!Species,
names_to = "Variavel",
values_to = "Valores") |>
ggplot(aes(x = Species, y = Valores)) +
geom_boxplot() +
facet_wrap(~ Variavel, scales = "free_y")
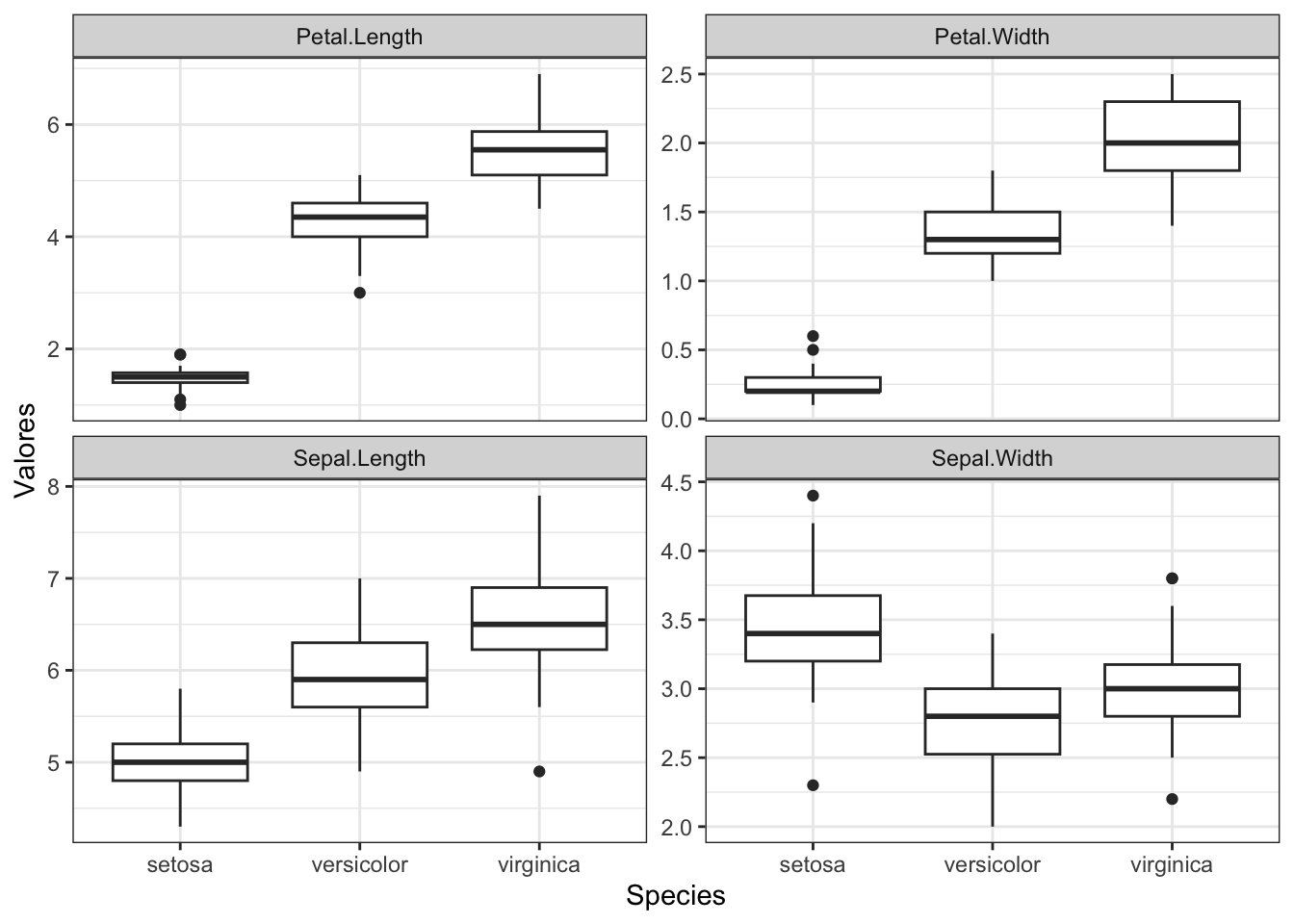
Muito mais fácil e prático, além de ser menos sujeito a erros e retrabalhos.