Introdução Link para o cabeçalho
Não deve ser surpresa para ninguém as reportagens sobre prováveis subnotificações a respeito dos casos de COVID-19. A fim de verificar se essas suspeitas são mesmo procedentes, eu decidi comparar os números de casos de Síndrome Respiratória Aguda Grave (SRAG) registrados no Brasil nos últimos 10 anos, a fim de tentar detectar se houve um aumento nos casos em 2020.
Para me ajudar nisso, vou usar um script do usuário belisards do github. O script se chama infogripe_scrap.R e ele baixa dados da SRAG coletados, organizados e disponibilizados pela Fiocruz no site Situação da Gripe. Para obter esses dados, basta rodar o script do belisards ou utilizar esse arquivo com os dados que já baixei até 16 de abril de 2020. Há um arquivo extra, com os dados por UF, mas ele não será utilizado nesse post.
Análise Exploratória Link para o cabeçalho
O primeiro passo é importar os dados para dentro do R.
library(tidyverse)
## ── Attaching core tidyverse packages ──────────────────────── tidyverse 2.0.0 ──
## ✔ dplyr 1.1.4 ✔ readr 2.1.5
## ✔ forcats 1.0.0 ✔ stringr 1.5.1
## ✔ ggplot2 3.5.1 ✔ tibble 3.2.1
## ✔ lubridate 1.9.3 ✔ tidyr 1.3.1
## ✔ purrr 1.0.2
## ── Conflicts ────────────────────────────────────────── tidyverse_conflicts() ──
## ✖ dplyr::filter() masks stats::filter()
## ✖ dplyr::lag() masks stats::lag()
## ℹ Use the conflicted package (<http://conflicted.r-lib.org/>) to force all conflicts to become errors
theme_set(theme_bw())
library(reshape2)
##
## Attaching package: 'reshape2'
##
## The following object is masked from 'package:tidyr':
##
## smiths
srag <- read_csv(file = "dados_br.csv")
## Rows: 586 Columns: 6
## ── Column specification ────────────────────────────────────────────────────────
## Delimiter: ","
## chr (3): territory_name, situation_name, value
## dbl (3): ano, epiweek, casos
##
## ℹ Use `spec()` to retrieve the full column specification for this data.
## ℹ Specify the column types or set `show_col_types = FALSE` to quiet this message.
Em seguida faço uma filtragem, mantendo apenas os últimos 10 anos na análise.
srag_filtrado <- srag %>%
filter(epiweek <= 14) %>%
filter(ano >= 2011) %>%
mutate(ano = factor(ano))
Por fim, faço as curvas com o número de casos de SRAG para cada ano de 2011 a 2020.
ggplot(srag_filtrado, aes(x = epiweek, y = casos, group = ano, colour = ano)) +
geom_line() +
scale_colour_viridis_d() +
scale_x_continuous(breaks = 1:14, labels = 1:14, minor_breaks = NULL) +
labs(x = "Semana", y = "Número de Casos", colour = "Ano",
title="Número de Casos de Síndrome Respiratória Aguda Grave por Ano")
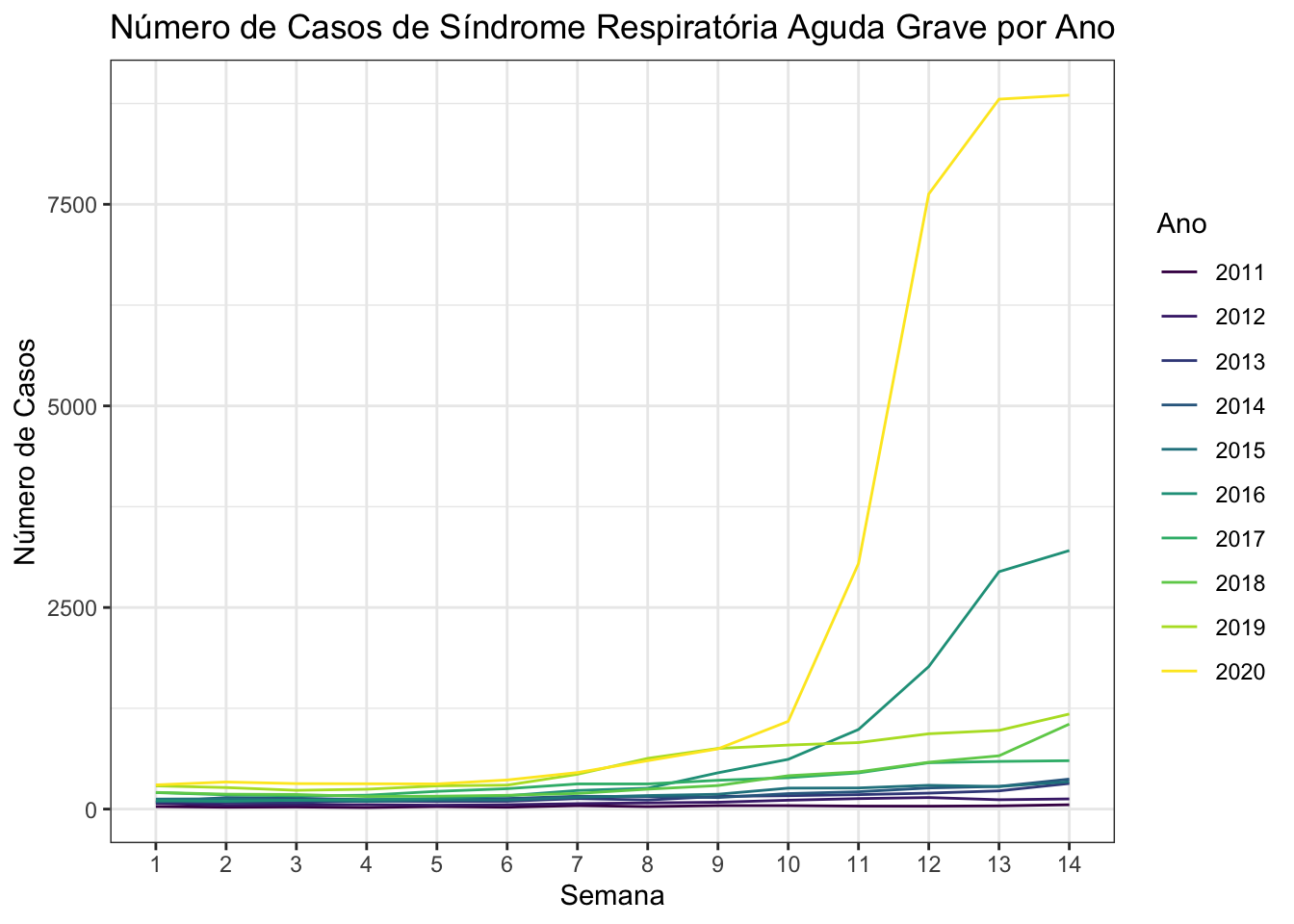
São dois os destaques que podemos ver no gráfico acima. O primeiro deles é o ano de 2020. Claramente está ocorrendo algum fenômeno que fez as notificações de SRAG aumentarem nesse ano, mas eu não faço ideia do que pode ser ¯_(ツ)_/¯
O segundo destaque é o ano de 2016. Eu não lembrava, mas parece que 2016 foi um ano com muitos casos dessa doença, com um número de mortes comparável à época pré-vacina. Atualização: segundo Leonardo Bastos, pesquisador da Fiocruz,
Sobre 2016 rolou uma grande epidemia (pouco reportda) de Influenza A (H1N1) que pegou bastante todo mundo de forma geral, mas em particular criancas de 0 a 2 anos.
No gráfico abaixo eu comparo o número de casos de SRAG em 2020 com a média histórica entre os anos de 2011 a 2019. A diferença fica ainda mais gritante.
srag_medio <- srag_filtrado %>%
filter(ano != "2020") %>%
group_by(epiweek) %>%
summarise(media = mean(casos))
full_join(srag_medio, filter(srag_filtrado, ano == "2020")) %>%
select(epiweek, media, casos) %>%
melt(id.var="epiweek") %>%
mutate(variable = ifelse(variable == "media", "Média\n2011-2019", "2020")) %>%
ggplot(., aes(x = epiweek, y = value, group = variable, colour = variable)) +
geom_line() +
scale_colour_viridis_d() +
scale_x_continuous(breaks = 1:14, labels = 1:14, minor_breaks = NULL) +
labs(x = "Semana", y = "Número de Casos", colour = "Grupo",
title="Número de Casos de Síndrome Respiratória Aguda Grave")
## Joining with `by = join_by(epiweek)`
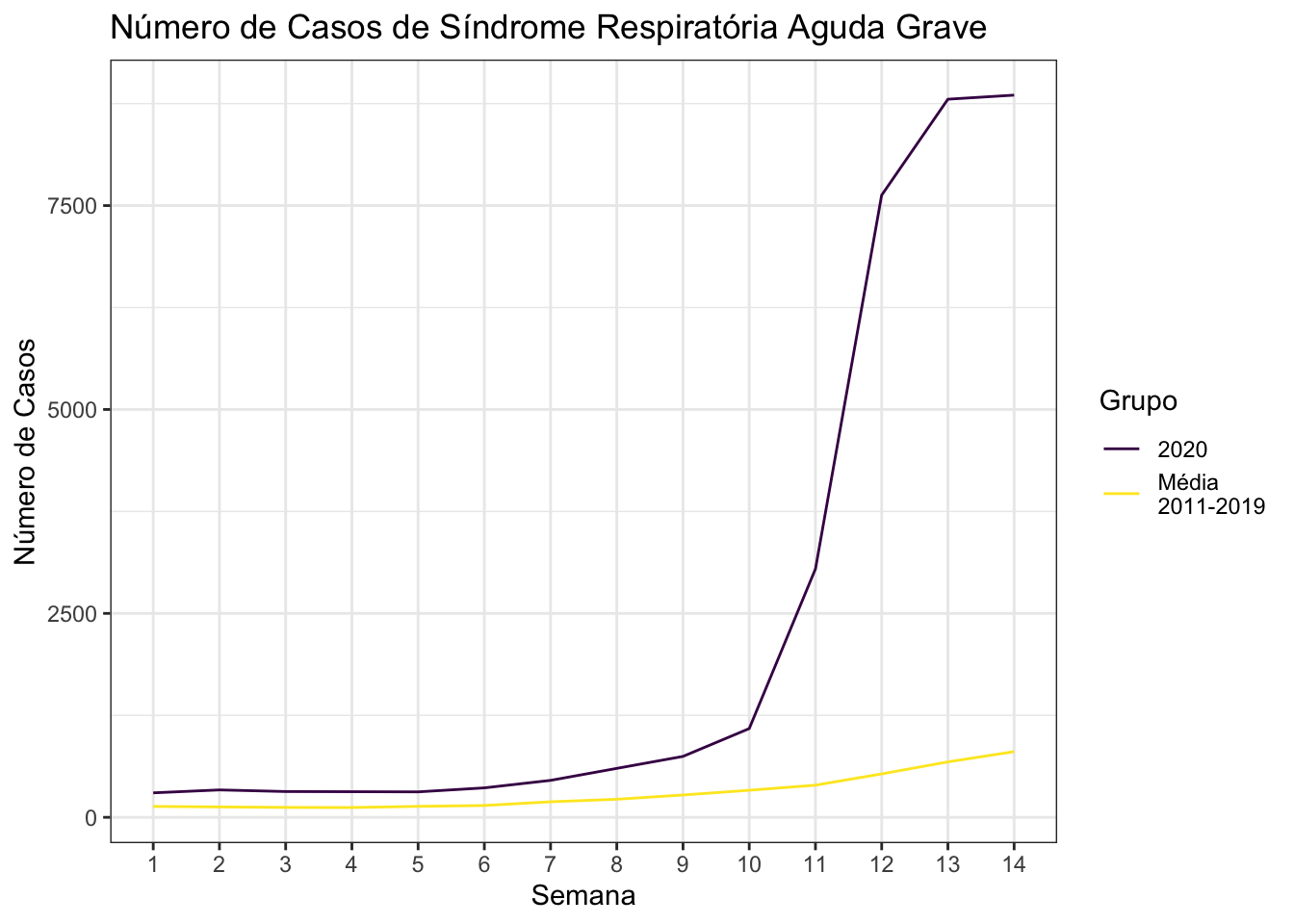
Creio que sequer seja necessário um teste de hipóteses para concluir que essas duas curvas são diferentes entre si.
Veja o post Dashboard: COVID-19 ou SRAG? para ver como construir um dashboard com os dados dos estados brasileiros.