Introdução Link para o cabeçalho
O paradoxo de Simpson aparece quando temos um conjunto de dados com uma tendência bem definida quando considerado completo, mas que muda completamente quando é separado em grupos.
Visualização do Paradoxo de Simpson Link para o cabeçalho
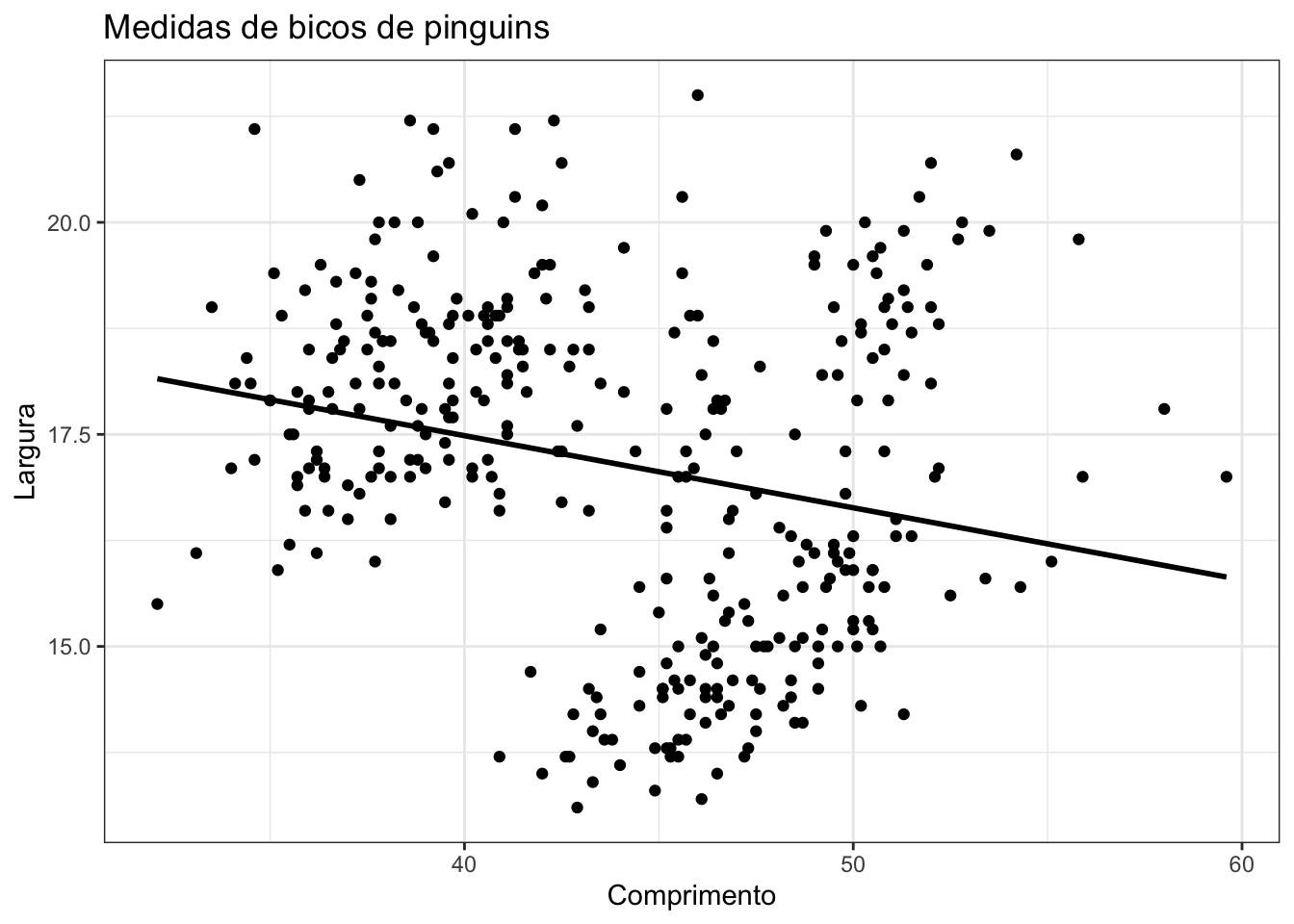
A melhor maneira de entender o que é o paradoxo de Simpson é visualizando-o na prática. A Figura a seguir mostra um gráfico de dispersão entre comprimento e largura de bicos de pinguins.
library(tidyverse)
## ── Attaching core tidyverse packages ──────────────────────── tidyverse 2.0.0 ──
## ✔ dplyr 1.1.4 ✔ readr 2.1.5
## ✔ forcats 1.0.0 ✔ stringr 1.5.1
## ✔ ggplot2 3.5.0 ✔ tibble 3.2.1
## ✔ lubridate 1.9.3 ✔ tidyr 1.3.1
## ✔ purrr 1.0.2
## ── Conflicts ────────────────────────────────────────── tidyverse_conflicts() ──
## ✖ dplyr::filter() masks stats::filter()
## ✖ dplyr::lag() masks stats::lag()
## ℹ Use the conflicted package (<http://conflicted.r-lib.org/>) to force all conflicts to become errors
theme_set(theme_bw())
library(broom)
library(palmerpenguins)
ggplot(penguins, aes(x = bill_length_mm, y = bill_depth_mm)) +
geom_point() +
geom_smooth(method = "lm", se = FALSE, colour = "black") +
labs(x = "Comprimento", y = "Largura", title = "Medidas de bicos de pinguins")
## `geom_smooth()` using formula = 'y ~ x'
penguins %>%
do(tidy(lm(bill_depth_mm ~ bill_length_mm, .)))
## # A tibble: 2 × 5
## term estimate std.error statistic p.value
## <chr> <dbl> <dbl> <dbl> <dbl>
## 1 (Intercept) 20.9 0.844 24.7 4.72e-78
## 2 bill_length_mm -0.0850 0.0191 -4.46 1.12e- 5
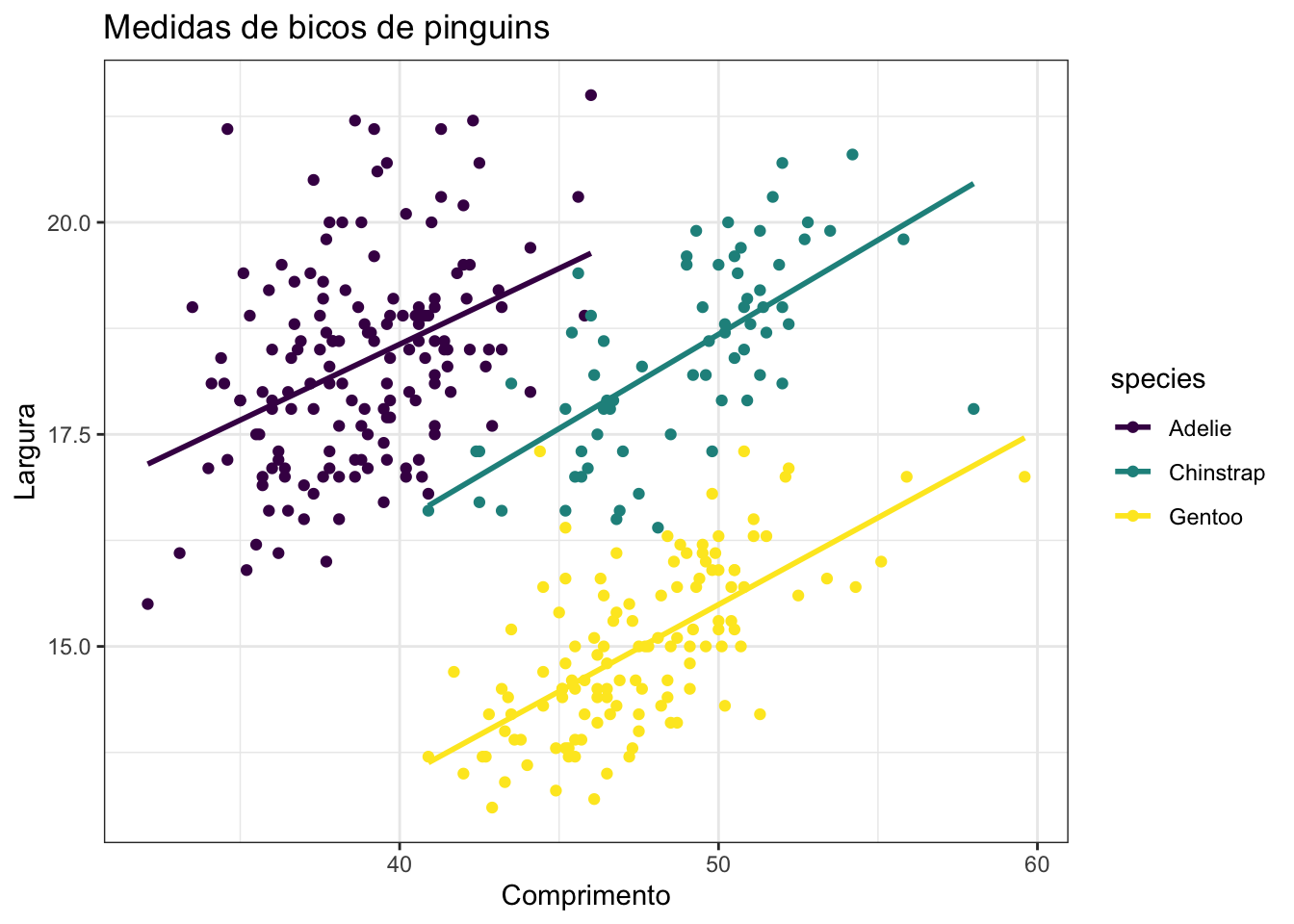
Como é possível ver acima, tanto gráfica quanto analiticamente, o coeficiente da regressão ajustada é negativo. Portanto, quanto mais comprido for o bico, menos largo ele será. Mas se criarmos uma regressão diferente para cada espécie de pinguim, o resultado obtido é diferente:
ggplot(penguins,
aes(x=bill_length_mm,
y=bill_depth_mm,
group = species,
colour = species)) +
geom_point() +
geom_smooth(aes(colour = species), method = "lm", se = FALSE) +
labs(x = "Comprimento", y = "Largura", title = "Medidas de bicos de pinguins", species = "Espécies") +
scale_colour_viridis_d()
## `geom_smooth()` using formula = 'y ~ x'
penguins %>%
group_by(species) %>%
do(tidy(lm(bill_depth_mm ~ bill_length_mm, .)))
## # A tibble: 6 × 6
## # Groups: species [3]
## species term estimate std.error statistic p.value
## <fct> <chr> <dbl> <dbl> <dbl> <dbl>
## 1 Adelie (Intercept) 11.4 1.34 8.52 1.61e-14
## 2 Adelie bill_length_mm 0.179 0.0344 5.19 6.67e- 7
## 3 Chinstrap (Intercept) 7.57 1.55 4.88 6.99e- 6
## 4 Chinstrap bill_length_mm 0.222 0.0317 7.01 1.53e- 9
## 5 Gentoo (Intercept) 5.25 1.05 4.98 2.15e- 6
## 6 Gentoo bill_length_mm 0.205 0.0222 9.24 1.02e-15
Perceba como as inclinações das retas se tornaram positivas, fato corroborado pelas equações das retas ajustadas a cada grupo.
Os códigos usados neste post podem ser baixados no meu github.